+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1659 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Solution structure and characterisation of the human pyruvate dehydrogenase complex core assembly | |||||||||
 マップデータ マップデータ | This contour level displays the E2-E3BP-E3 structure with ordered extensions from the core surface and some noisier density not following the icosahedral symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pyruvate dehydrogenase complex / dodecahedron | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 33.0 Å | |||||||||
 データ登録者 データ登録者 | Vijayakrishnan S / Kelly SM / Gilbert RJC / Callow P / Bhella D / Forsyth T / Lindsay JG / Byron O | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2010 ジャーナル: J Mol Biol / 年: 2010タイトル: Solution structure and characterisation of the human pyruvate dehydrogenase complex core assembly. 著者: S Vijayakrishnan / S M Kelly / R J C Gilbert / P Callow / D Bhella / T Forsyth / J G Lindsay / O Byron /  要旨: Mammalian pyruvate dehydrogenase complex (PDC) is a key multi-enzyme assembly that is responsible for glucose homeostasis maintenance and conversion of pyruvate into acetyl-CoA. It comprises a ...Mammalian pyruvate dehydrogenase complex (PDC) is a key multi-enzyme assembly that is responsible for glucose homeostasis maintenance and conversion of pyruvate into acetyl-CoA. It comprises a central pentagonal dodecahedral core consisting of two subunit types (E2 and E3BP) to which peripheral enzymes (E1 and E3) bind tightly but non-covalently. Currently, there are two conflicting models of PDC (E2+E3BP) core organisation: the 'addition' model (60+12) and the 'substitution' model (48+12). Here we present the first ever low-resolution structures of human recombinant full-length PDC core (rE2/E3BP), truncated PDC core (tE2/E3BP) and native bovine heart PDC core (bE2/E3BP) obtained by small-angle X-ray scattering and small-angle neutron scattering. These structures, corroborated by negative-stain and cryo electron microscopy data, clearly reveal open pentagonal core faces, favouring the 'substitution' model of core organisation. The native and recombinant core structures are all similar to the truncated bacterial E2 core crystal structure obtained previously. Cryo-electron microscopy reconstructions of rE2/E3BP and rE2/E3BP:E3 directly confirm that the core has open pentagonal faces, agree with scattering-derived models and show density extending outwards from their surfaces, which is much more structurally ordered in the presence of E3. Additionally, analytical ultracentrifugation characterisation of rE2/E3BP, rE2 (full-length recombinant E2-only) and tE2/E3BP supports the substitution model. Superimposition of the small-angle neutron scattering tE2/E3BP and truncated bacterial E2 crystal structures demonstrates conservation of the overall pentagonal dodecahedral morphology, despite evolutionary diversity. In addition, unfolding studies using circular dichroism and tryptophan fluorescence spectroscopy show that the rE2/E3BP is less stable than its rE2 counterpart, indicative of a role for E3BP in core destabilisation. The architectural complexity and lower stability of the E2/E3BP core may be of benefit to mammals, where sophisticated fine-tuning is required for cores with optimal catalytic and regulatory efficiencies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1659.map.gz emd_1659.map.gz | 28.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1659-v30.xml emd-1659-v30.xml emd-1659.xml emd-1659.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1659.gif EMD-1659.gif | 129.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1659 http://ftp.pdbj.org/pub/emdb/structures/EMD-1659 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1659 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1659 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1659_validation.pdf.gz emd_1659_validation.pdf.gz | 240.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1659_full_validation.pdf.gz emd_1659_full_validation.pdf.gz | 239.6 KB | 表示 | |
| XML形式データ |  emd_1659_validation.xml.gz emd_1659_validation.xml.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1659 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1659 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1659 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1659 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1659.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1659.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This contour level displays the E2-E3BP-E3 structure with ordered extensions from the core surface and some noisier density not following the icosahedral symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
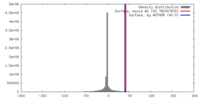
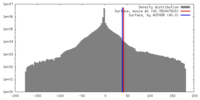
| ボクセルのサイズ | X=Y=Z: 3.6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pyruvate dehydrogenase E2-E3BP-E3 complex
| 全体 | 名称: Pyruvate dehydrogenase E2-E3BP-E3 complex |
|---|---|
| 要素 |
|
-超分子 #1000: Pyruvate dehydrogenase E2-E3BP-E3 complex
| 超分子 | 名称: Pyruvate dehydrogenase E2-E3BP-E3 complex / タイプ: sample / ID: 1000 集合状態: A dodecahedral lattice formed of mixed trimers of E2 and E3BP, most likely each containing 2 E2 and 1 E3BP, bound with the E3 Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 5.4 MDa |
-分子 #1: dihydrolipoamide dehydrogenase
| 分子 | 名称: dihydrolipoamide dehydrogenase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: E3 / コピー数: 20 / 集合状態: Hetero-icosahedron / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 60 KDa |
| 組換発現 | 生物種:  |
-分子 #2: E3 binding protein
| 分子 | 名称: E3 binding protein / タイプ: protein_or_peptide / ID: 2 / Name.synonym: E3BP / コピー数: 20 / 集合状態: Hetero-icosahedron / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 60 KDa |
| 組換発現 | 生物種:  |
-分子 #3: Dihydrolipoyl transacetylase
| 分子 | 名称: Dihydrolipoyl transacetylase / タイプ: protein_or_peptide / ID: 3 / Name.synonym: E2 / コピー数: 40 / 集合状態: Hetero-icosahedron / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 70 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 2 mM EDTA, 0.01% (w/v) NaN3, 100 mM NaCl, 50 mM KH2PO4, pH 7.5 |
|---|---|
| グリッド | 詳細: 300 mesh copper grid with lacey carbon film |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 100 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Homemade plunger 手法: Blot briefly before plunging, using Whatman no.1 paper |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 平均: 115 K |
| アライメント法 | Legacy - 非点収差: At 200,000 magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 9 詳細: Images were binned from a scanned pixel size at the specimen of 1.8A to 3.6A using Proc2D Od range: 5 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles selected interactively using Boxer |
|---|---|
| CTF補正 | 詳細: Per micrograph |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 33.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider, WellMAP 詳細: Final map had converged to given resolution, on a 1 degree spacing. 使用した粒子像数: 172 |
| 最終 角度割当 | 詳細: SPIDER convention |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | PDBEntryID_givenInChain. Protocol: Rigid body, after icosahedral symmetrization. The icosahedral assembly was constructed and then fitted as a rigid body. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CCC |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)