+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the Hendra henipavirus nucleocapsid sauronoid assembly multimer | |||||||||
 マップデータ マップデータ | Hendra henipavirus nucleoprotein sauronoid EM density postprocessed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleoprotein / RNA-binding protein / Sauronoid / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Paramyxovirus nucleocapsid protein / Paramyxovirus nucleocapsid protein / helical viral capsid / viral nucleocapsid / host cell cytoplasm / ribonucleoprotein complex / structural molecule activity / RNA binding / Nucleocapsid 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Hendra henipavirus (ウイルス) / Hendra henipavirus (ウイルス) /  | |||||||||
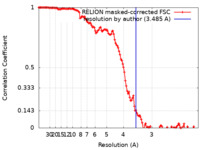
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.485 Å | |||||||||
 データ登録者 データ登録者 | Passchier TC / Maskell DP / Edwards TA / Barr JN | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2024 ジャーナル: Sci Rep / 年: 2024タイトル: The cryoEM structure of the Hendra henipavirus nucleoprotein reveals insights into paramyxoviral nucleocapsid architectures. 著者: Tim C Passchier / Joshua B R White / Daniel P Maskell / Matthew J Byrne / Neil A Ranson / Thomas A Edwards / John N Barr /   要旨: We report the first cryoEM structure of the Hendra henipavirus nucleoprotein in complex with RNA, at 3.5 Å resolution, derived from single particle analysis of a double homotetradecameric RNA-bound ...We report the first cryoEM structure of the Hendra henipavirus nucleoprotein in complex with RNA, at 3.5 Å resolution, derived from single particle analysis of a double homotetradecameric RNA-bound N protein ring assembly exhibiting D14 symmetry. The structure of the HeV N protein adopts the common bi-lobed paramyxoviral N protein fold; the N-terminal and C-terminal globular domains are bisected by an RNA binding cleft containing six RNA nucleotides and are flanked by the N-terminal and C-terminal arms, respectively. In common with other paramyxoviral nucleocapsids, the lateral interface between adjacent N and N protomers involves electrostatic and hydrophobic interactions mediated primarily through the N-terminal arm and globular domains with minor contribution from the C-terminal arm. However, the HeV N multimeric assembly uniquely identifies an additional protomer-protomer contact between the N N-terminus and N C-terminal arm linker. The model presented here broadens the understanding of RNA-bound paramyxoviral nucleocapsid architectures and provides a platform for further insight into the molecular biology of HeV, as well as the development of antiviral interventions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16426.map.gz emd_16426.map.gz | 16.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16426-v30.xml emd-16426-v30.xml emd-16426.xml emd-16426.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16426_fsc.xml emd_16426_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16426.png emd_16426.png | 117.6 KB | ||
| マスクデータ |  emd_16426_msk_1.map emd_16426_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16426.cif.gz emd-16426.cif.gz | 6.2 KB | ||
| その他 |  emd_16426_half_map_1.map.gz emd_16426_half_map_1.map.gz emd_16426_half_map_2.map.gz emd_16426_half_map_2.map.gz | 138.3 MB 138.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16426 http://ftp.pdbj.org/pub/emdb/structures/EMD-16426 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16426 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16426 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16426_validation.pdf.gz emd_16426_validation.pdf.gz | 848.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16426_full_validation.pdf.gz emd_16426_full_validation.pdf.gz | 848.4 KB | 表示 | |
| XML形式データ |  emd_16426_validation.xml.gz emd_16426_validation.xml.gz | 20.3 KB | 表示 | |
| CIF形式データ |  emd_16426_validation.cif.gz emd_16426_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16426 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16426 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16426 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16426 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c4hMC  8cbwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16426.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16426.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hendra henipavirus nucleoprotein sauronoid EM density postprocessed map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||
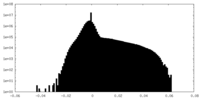
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16426_msk_1.map emd_16426_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
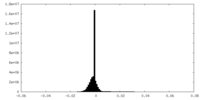
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Hendra henipavirus nucleoprotein sauronoid EM density half map 2
| ファイル | emd_16426_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hendra henipavirus nucleoprotein sauronoid EM density half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Hendra henipavirus nucleoprotein sauronoid EM density half map 1
| ファイル | emd_16426_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hendra henipavirus nucleoprotein sauronoid EM density half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sauronoid assembly of Hendra henipavirus nucleoprotein
| 全体 | 名称: Sauronoid assembly of Hendra henipavirus nucleoprotein |
|---|---|
| 要素 |
|
-超分子 #1: Sauronoid assembly of Hendra henipavirus nucleoprotein
| 超分子 | 名称: Sauronoid assembly of Hendra henipavirus nucleoprotein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Recombinantly expressed Hendra henipavirus nucleoprotein forming sauronoid complexes. |
|---|---|
| 由来(天然) | 生物種:  Hendra henipavirus (ウイルス) Hendra henipavirus (ウイルス) |
| 分子量 | 理論値: 1.691779 MDa |
-分子 #1: Nucleocapsid
| 分子 | 名称: Nucleocapsid / タイプ: protein_or_peptide / ID: 1 / コピー数: 28 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hendra henipavirus (ウイルス) Hendra henipavirus (ウイルス) |
| 分子量 | 理論値: 58.54132 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDIFDEAAS FRSYQSKLGR DGRASAATAT LTTKIRIFVP ATNSPELRWE LTLFALDVIR SPSAAESMKI GAAFTLISMY SERPGALIR SLLNDPDIEA VIIDVGSMLN GIPVMERRGD KAQEEMEGLM RILKTARESS KGKTPFVDSR AYGLRITDMS T LVSAVITI ...文字列: MSDIFDEAAS FRSYQSKLGR DGRASAATAT LTTKIRIFVP ATNSPELRWE LTLFALDVIR SPSAAESMKI GAAFTLISMY SERPGALIR SLLNDPDIEA VIIDVGSMLN GIPVMERRGD KAQEEMEGLM RILKTARESS KGKTPFVDSR AYGLRITDMS T LVSAVITI EAQIWILIAK AVTAPDTAEE SETRRWAKYV QQKRVNPFFA LTQQWLTEMR NLLSQSLSVR KFMVEILMEV KK GGSAKGR AVEIISDIGN YVEETGMAGF FATIRFGLET RYPALALNEF QSDLNTIKGL MLLYREIGPR APYMVLLEES IQT KFAPGG YPLLWSFAMG VATTIDRSMG ALNINRGYLE PMYFRLGQKS ARHHAGGIDQ NMANKLGLNS DQVAELAAAV QETS VGRQD NNMQAREAKF AAGGVLVGGG EQDIDEEEEP IEHSGRQSVT FKREMSMSSL ADSVPSSSVS TSGGTRLTNS LLNLR SRLA AKAIKESTAQ SSSERNPPNN RPQADSGRKD DQEPKPAQND LDFVRADV UniProtKB: Nucleocapsid |
-分子 #2: RNA (84-MER)
| 分子 | 名称: RNA (84-MER) / タイプ: rna / ID: 2 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.672984 KDa |
| 配列 | 文字列: UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUUUUUUUU UUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 3548 / 平均露光時間: 1.5 sec. / 平均電子線量: 59.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X