+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Urea Transporter UT-B/UT1 in Complex with Inhibitor UTBinh-14 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SLC14A1 / UT1 / UT-B / Urea Transporter / Inhibitor / Solute Carrier / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報urea channel activity / urea transport / Transport of bile salts and organic acids, metal ions and amine compounds / urea transmembrane transport / urea transmembrane transporter activity / water transmembrane transporter activity / establishment of localization in cell / transmembrane transport / basolateral plasma membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Chi G / Dietz L / Pike ACW / Maclean EM / Mukhopadhyay SMM / Bohstedt T / Wang D / Scacioc A / McKinley G / Arrowsmith CH ...Chi G / Dietz L / Pike ACW / Maclean EM / Mukhopadhyay SMM / Bohstedt T / Wang D / Scacioc A / McKinley G / Arrowsmith CH / Edwards A / Bountra C / Fernandez-Cid A / Burgess-Brown NA / Duerr KL | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Structural characterization of human urea transporters UT-A and UT-B and their inhibition. 著者: Gamma Chi / Larissa Dietz / Haiping Tang / Matthew Snee / Andreea Scacioc / Dong Wang / Gavin Mckinley / Shubhashish M M Mukhopadhyay / Ashley C W Pike / Rod Chalk / Nicola A Burgess-Brown / ...著者: Gamma Chi / Larissa Dietz / Haiping Tang / Matthew Snee / Andreea Scacioc / Dong Wang / Gavin Mckinley / Shubhashish M M Mukhopadhyay / Ashley C W Pike / Rod Chalk / Nicola A Burgess-Brown / Jean-Pierre Timmermans / Wouter van Putte / Carol V Robinson / Katharina L Dürr /   要旨: In this study, we present the structures of human urea transporters UT-A and UT-B to characterize them at molecular level and to detail the mechanism of UT-B inhibition by its selective inhibitor, ...In this study, we present the structures of human urea transporters UT-A and UT-B to characterize them at molecular level and to detail the mechanism of UT-B inhibition by its selective inhibitor, UTB-14. High-resolution structures of both transporters establish the structural basis for the inhibitor's selectivity to UT-B, and the identification of multiple binding sites for the inhibitor will aid with the development of drug lead molecules targeting both transporters. Our study also discovers phospholipids associating with the urea transporters by combining structural observations, native MS, and lipidomics analysis. These insights improve our understanding of urea transporter function at a molecular level and provide a blueprint for a structure-guided design of therapeutics targeting these transporters. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16112.map.gz emd_16112.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16112-v30.xml emd-16112-v30.xml emd-16112.xml emd-16112.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16112_fsc.xml emd_16112_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16112.png emd_16112.png | 201.5 KB | ||
| マスクデータ |  emd_16112_msk_1.map emd_16112_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16112.cif.gz emd-16112.cif.gz | 6.2 KB | ||
| その他 |  emd_16112_half_map_1.map.gz emd_16112_half_map_1.map.gz emd_16112_half_map_2.map.gz emd_16112_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16112 http://ftp.pdbj.org/pub/emdb/structures/EMD-16112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16112 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16112.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16112.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16112_msk_1.map emd_16112_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
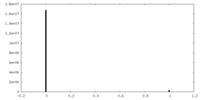
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16112_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
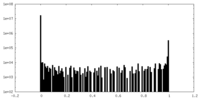
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16112_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homotrimeric complex of Human Urea Transporter UT-B
| 全体 | 名称: Homotrimeric complex of Human Urea Transporter UT-B |
|---|---|
| 要素 |
|
-超分子 #1: Homotrimeric complex of Human Urea Transporter UT-B
| 超分子 | 名称: Homotrimeric complex of Human Urea Transporter UT-B / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Urea transporter 1
| 分子 | 名称: Urea transporter 1 / タイプ: protein_or_peptide / ID: 1 詳細: N211A is an engineered mutation. The rest are natural variants. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.17741 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFPKALGYVT GDMKELANQL KDKPVVLQFI DWILRGISQV VFVNNPVSGI LILVGLLVQN PWWALTGWLG TVVSTLMALL LSQDRSLIA SGLYGYNATL VGVLMAVFSD KGDYFWWLLL PVCAMSMTCP IFSSALNSVL SKWDLPVFTL PFNMALSMYL S ATGHYNPF ...文字列: MFPKALGYVT GDMKELANQL KDKPVVLQFI DWILRGISQV VFVNNPVSGI LILVGLLVQN PWWALTGWLG TVVSTLMALL LSQDRSLIA SGLYGYNATL VGVLMAVFSD KGDYFWWLLL PVCAMSMTCP IFSSALNSVL SKWDLPVFTL PFNMALSMYL S ATGHYNPF FPAKLVIPIT TAPQISWSDL SALELLKSIP VGVGQIYGCD NPWTGGIFLG AILLSSPLMC LHAAIGSLLG IA AGLSLSA PFENIYFGLW GFNSSLACIA MGGMFMALTW QTHLLALGCA LFTAYLGVGM ANFMAEVGLP ACTWPFCLAT LLF LIMTTK NSNIYKMPLS KVTYPEENRI FYLQAKKRMV ESPLAENLYF Q UniProtKB: Urea transporter 1 |
-分子 #2: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #3: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 3 / コピー数: 51 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #4: 10-(4-ethylphenyl)sulfonyl-~{N}-(thiophen-2-ylmethyl)-5-thia-1,8,...
| 分子 | 名称: 10-(4-ethylphenyl)sulfonyl-~{N}-(thiophen-2-ylmethyl)-5-thia-1,8,11,12-tetrazatricyclo[7.3.0.0^{2,6}]dodeca-2(6),3,7,9,11-pentaen-7-amine タイプ: ligand / ID: 4 / コピー数: 6 / 式: 5D3 |
|---|---|
| 分子量 | 理論値: 455.576 Da |
| Chemical component information |  ChemComp-5D3: |
-分子 #5: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 66 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 8221 / 平均電子線量: 39.58 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8blp: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X