+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of beta-galactosidase at 3.3 A resolution plunged 5 ms after mixing with apoferritin | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Glycosyl hydrolase / HYDROLASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alkali metal ion binding / lactose catabolic process / beta-galactosidase complex / beta-galactosidase / beta-galactosidase activity / carbohydrate binding / magnesium ion binding / identical protein binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   | ||||||||||||
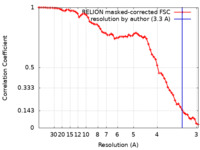
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Torino S / Dhurandhar M / Efremov R | ||||||||||||
| 資金援助 | European Union,  ベルギー, 3件 ベルギー, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2023 ジャーナル: Nat Methods / 年: 2023タイトル: Time-resolved cryo-EM using a combination of droplet microfluidics with on-demand jetting. 著者: Stefania Torino / Mugdha Dhurandhar / Annelore Stroobants / Raf Claessens / Rouslan G Efremov /  要旨: Single-particle cryogenic electron microscopy (cryo-EM) allows reconstruction of high-resolution structures of proteins in different conformations. Protein function often involves transient ...Single-particle cryogenic electron microscopy (cryo-EM) allows reconstruction of high-resolution structures of proteins in different conformations. Protein function often involves transient functional conformations, which can be resolved using time-resolved cryo-EM (trEM). In trEM, reactions are arrested after a defined delay time by rapid vitrification of protein solution on the EM grid. Despite the increasing interest in trEM among the cryo-EM community, making trEM samples with a time resolution below 100 ms remains challenging. Here we report the design and the realization of a time-resolved cryo-plunger that combines a droplet-based microfluidic mixer with a laser-induced generator of microjets that allows rapid reaction initiation and plunge-freezing of cryo-EM grids. Using this approach, a time resolution of 5 ms was achieved and the protein density map was reconstructed to a resolution of 2.1 Å. trEM experiments on GroEL:GroES chaperonin complex resolved the kinetics of the complex formation and visualized putative short-lived conformations of GroEL-ATP complex. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16091.map.gz emd_16091.map.gz | 20.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16091-v30.xml emd-16091-v30.xml emd-16091.xml emd-16091.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16091_fsc.xml emd_16091_fsc.xml | 6.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16091.png emd_16091.png | 150.4 KB | ||
| マスクデータ |  emd_16091_msk_1.map emd_16091_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16091_half_map_1.map.gz emd_16091_half_map_1.map.gz emd_16091_half_map_2.map.gz emd_16091_half_map_2.map.gz | 16.1 MB 16.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16091 http://ftp.pdbj.org/pub/emdb/structures/EMD-16091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16091 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16091_validation.pdf.gz emd_16091_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16091_full_validation.pdf.gz emd_16091_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_16091_validation.xml.gz emd_16091_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_16091_validation.cif.gz emd_16091_validation.cif.gz | 16.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16091 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16091 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bk7MC  8bk8C  8bk9C  8bkaC  8bkbC  8bkgC  8bkzC  8bl2C  8bl7C  8blcC  8bldC  8bleC  8blfC  8blyC  8bm0C  8bm1C  8bmdC  8bmoC  8bmtC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16091.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16091.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.486 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16091_msk_1.map emd_16091_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16091_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16091_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Beta-galactosidase from E.Coli
| 全体 | 名称: Beta-galactosidase from E.Coli |
|---|---|
| 要素 |
|
-超分子 #1: Beta-galactosidase from E.Coli
| 超分子 | 名称: Beta-galactosidase from E.Coli / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Beta-galactosidase
| 分子 | 名称: Beta-galactosidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: beta-galactosidase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 117.488375 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTMITDSLAV VLQRRDWENP GVTQLNRLAA HPPFASWRNS EEARTDRPSQ QLRSLNGEWR FAWFPAPEAV PESWLECDLP EADTVVVPS NWQMHGYDAP IYTNVTYPIT VNPPFVPTEN PTGCYSLTFN VDESWLQEGQ TRIIFDGVNS AFHLWCNGRW V GYGQDSRL ...文字列: MTMITDSLAV VLQRRDWENP GVTQLNRLAA HPPFASWRNS EEARTDRPSQ QLRSLNGEWR FAWFPAPEAV PESWLECDLP EADTVVVPS NWQMHGYDAP IYTNVTYPIT VNPPFVPTEN PTGCYSLTFN VDESWLQEGQ TRIIFDGVNS AFHLWCNGRW V GYGQDSRL PSEFDLSAFL RAGENRLAVM VLRWSDGSYL EDQDMWRMSG IFRDVSLLHK PTTQISDFHV ATRFNDDFSR AV LEAEVQM CGELRDYLRV TVSLWQGETQ VASGTAPFGG EIIDERGGYA DRVTLRLNVE NPKLWSAEIP NLYRAVVELH TAD GTLIEA EACDVGFREV RIENGLLLLN GKPLLIRGVN RHEHHPLHGQ VMDEQTMVQD ILLMKQNNFN AVRCSHYPNH PLWY TLCDR YGLYVVDEAN IETHGMVPMN RLTDDPRWLP AMSERVTRMV QRDRNHPSVI IWSLGNESGH GANHDALYRW IKSVD PSRP VQYEGGGADT TATDIICPMY ARVDEDQPFP AVPKWSIKKW LSLPGETRPL ILCEYAHAMG NSLGGFAKYW QAFRQY PRL QGGFVWDWVD QSLIKYDENG NPWSAYGGDF GDTPNDRQFC MNGLVFADRT PHPALTEAKH QQQFFQFRLS GQTIEVT SE YLFRHSDNEL LHWMVALDGK PLASGEVPLD VAPQGKQLIE LPELPQPESA GQLWLTVRVV QPNATAWSEA GHISAWQQ W RLAENLSVTL PAASHAIPHL TTSEMDFCIE LGNKRWQFNR QSGFLSQMWI GDKKQLLTPL RDQFTRAPLD NDIGVSEAT RIDPNAWVER WKAAGHYQAE AALLQCTADT LADAVLITTA HAWQHQGKTL FISRKTYRID GSGQMAITVD VEVASDTPHP ARIGLNCQL AQVAERVNWL GLGPQENYPD RLTAACFDRW DLPLSDMYTP YVFPSENGLR CGTRELNYGP HQWRGDFQFN I SRYSQQQL METSHRHLLH AEEGTWLNID GFHMGIGGDD SWSPSVSAEF QLSAGRYHYQ LVWCQKGHHH HHH UniProtKB: Beta-galactosidase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.7 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: contains Amaranth dye (acid red 27) 32 mM | |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE | |||||||||||||||
| 詳細 | Beta-galactosidase monodisperse in buffer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 1733 / 平均露光時間: 2.796 sec. / 平均電子線量: 59.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.55 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)