+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human full length RAD52 undecamer | |||||||||
 マップデータ マップデータ | Human full length RAD52 cryo-electron density map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA repair protein / oligomeric structure / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報double-strand break repair via single-strand annealing / DNA double-strand break processing involved in repair via single-strand annealing / DNA recombinase assembly / regulation of nucleotide-excision repair / mitotic recombination / HDR through MMEJ (alt-NHEJ) / HDR through Single Strand Annealing (SSA) / SUMOylation of DNA damage response and repair proteins / protein-DNA complex / double-strand break repair via homologous recombination ...double-strand break repair via single-strand annealing / DNA double-strand break processing involved in repair via single-strand annealing / DNA recombinase assembly / regulation of nucleotide-excision repair / mitotic recombination / HDR through MMEJ (alt-NHEJ) / HDR through Single Strand Annealing (SSA) / SUMOylation of DNA damage response and repair proteins / protein-DNA complex / double-strand break repair via homologous recombination / double-strand break repair / single-stranded DNA binding / cellular response to oxidative stress / DNA recombination / protein-containing complex / DNA binding / nucleoplasm / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
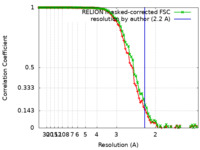
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Marotta R / Balboni B / Girotto S / Cavalli A | |||||||||
| 資金援助 | European Union,  イタリア, 2件 イタリア, 2件
| |||||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: Novel structural insights on full-length human RAD52: Cryo-EM and beyond 著者: Balboni B / Marotta R / Rinaldi F / Girotto S / Cavalli A | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16089.map.gz emd_16089.map.gz | 27.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16089-v30.xml emd-16089-v30.xml emd-16089.xml emd-16089.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16089_fsc.xml emd_16089_fsc.xml emd_16089_fsc_2.xml emd_16089_fsc_2.xml | 9.6 KB 7.5 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16089.png emd_16089.png | 34.9 KB | ||
| Filedesc metadata |  emd-16089.cif.gz emd-16089.cif.gz | 5.9 KB | ||
| その他 |  emd_16089_half_map_1.map.gz emd_16089_half_map_1.map.gz emd_16089_half_map_2.map.gz emd_16089_half_map_2.map.gz | 27.1 MB 27.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16089 http://ftp.pdbj.org/pub/emdb/structures/EMD-16089 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16089 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16089 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16089_validation.pdf.gz emd_16089_validation.pdf.gz | 787 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16089_full_validation.pdf.gz emd_16089_full_validation.pdf.gz | 786.6 KB | 表示 | |
| XML形式データ |  emd_16089_validation.xml.gz emd_16089_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_16089_validation.cif.gz emd_16089_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16089 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16089 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16089 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16089 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bjmMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16089.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16089.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human full length RAD52 cryo-electron density map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.731 Å | ||||||||||||||||||||||||||||||||||||
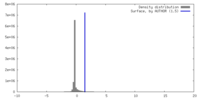
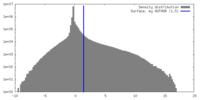
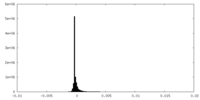
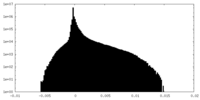
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Human full-length RAD52 half cryo-electron density map 1
| ファイル | emd_16089_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human full-length RAD52 half cryo-electron density map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Human full-length RAD52 half cryo-electron density map 2
| ファイル | emd_16089_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human full-length RAD52 half cryo-electron density map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human full length RAD52
| 全体 | 名称: Human full length RAD52 |
|---|---|
| 要素 |
|
-超分子 #1: Human full length RAD52
| 超分子 | 名称: Human full length RAD52 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: N-terminal 6xHis-RAD52 recombinant protein |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 650 KDa |
-分子 #1: DNA repair protein RAD52 homolog
| 分子 | 名称: DNA repair protein RAD52 homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 48.044637 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHENL YFQGMSGTEE AILGGRDSHP AAGGGSVLCF GQCQYTAEEY QAIQKALRQR LGPEYISSRM AGGGQKVCYI EGHRVINLA NEMFGYNGWA HSITQQNVDF VDLNNGKFYV GVCAFVRVQL KDGSYHEDVG YGVSEGLKSK ALSLEKARKE A VTDGLKRA ...文字列: MHHHHHHENL YFQGMSGTEE AILGGRDSHP AAGGGSVLCF GQCQYTAEEY QAIQKALRQR LGPEYISSRM AGGGQKVCYI EGHRVINLA NEMFGYNGWA HSITQQNVDF VDLNNGKFYV GVCAFVRVQL KDGSYHEDVG YGVSEGLKSK ALSLEKARKE A VTDGLKRA LRSFGNALGN CILDKDYLRS LNKLPRQLPL EVDLTKAKRQ DLEPSVEEAR YNSCRPNMAL GHPQLQQVTS PS RPSHAVI PADQDCSSRS LSSSAVESEA THQRKLRQKQ LQQQFRERME KQQVRVSTPS AEKSEAAPPA PPVTHSTPVT VSE PLLEKD FLAGVTQELI KTLEDNSEKW AVTPDAGDGV VKPSSRADPA QTSDTLALNN QMVTQNRTPH SVCHQKPQAK SGSW DLQTY SADQRTTGNW ESHRKSQDMK KRKYDPS UniProtKB: DNA repair protein RAD52 homolog |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 498 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 318.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 17400 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)