+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | IstA transposase cleaved donor complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA Transposition / Transposase / Cleaved donor complex / DDE domain / IS21 / IstA / Insertion sequence / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) | |||||||||
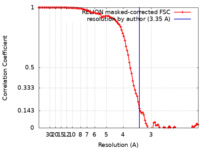
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Spinola-Amilibia M / de la Gandara A / Araujo-Bazan L / Berger JM / Arias-Palomo E | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: IS21 family transposase cleaved donor complex traps two right-handed superhelical crossings. 著者: Mercedes Spínola-Amilibia / Lidia Araújo-Bazán / Álvaro de la Gándara / James M Berger / Ernesto Arias-Palomo /   要旨: Transposases are ubiquitous enzymes that catalyze DNA rearrangement events with broad impacts on gene expression, genome evolution, and the spread of drug-resistance in bacteria. Here, we use ...Transposases are ubiquitous enzymes that catalyze DNA rearrangement events with broad impacts on gene expression, genome evolution, and the spread of drug-resistance in bacteria. Here, we use biochemical and structural approaches to define the molecular determinants by which IstA, a transposase present in the widespread IS21 family of mobile elements, catalyzes efficient DNA transposition. Solution studies show that IstA engages the transposon terminal sequences to form a high-molecular weight complex and promote DNA integration. A 3.4 Å resolution structure of the transposase bound to transposon ends corroborates our biochemical findings and reveals that IstA self-assembles into a highly intertwined tetramer that synapses two supercoiled terminal inverted repeats. The three-dimensional organization of the IstA•DNA cleaved donor complex reveals remarkable similarities with retroviral integrases and classic transposase systems, such as Tn7 and bacteriophage Mu, and provides insights into IS21 transposition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15848.map.gz emd_15848.map.gz | 85.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15848-v30.xml emd-15848-v30.xml emd-15848.xml emd-15848.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15848_fsc.xml emd_15848_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15848.png emd_15848.png | 72.8 KB | ||
| マスクデータ |  emd_15848_msk_1.map emd_15848_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15848.cif.gz emd-15848.cif.gz | 6.9 KB | ||
| その他 |  emd_15848_additional_1.map.gz emd_15848_additional_1.map.gz emd_15848_half_map_1.map.gz emd_15848_half_map_1.map.gz emd_15848_half_map_2.map.gz emd_15848_half_map_2.map.gz | 69.9 MB 70.1 MB 70.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15848 http://ftp.pdbj.org/pub/emdb/structures/EMD-15848 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15848 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15848 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15848_validation.pdf.gz emd_15848_validation.pdf.gz | 840 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15848_full_validation.pdf.gz emd_15848_full_validation.pdf.gz | 839.6 KB | 表示 | |
| XML形式データ |  emd_15848_validation.xml.gz emd_15848_validation.xml.gz | 17.1 KB | 表示 | |
| CIF形式データ |  emd_15848_validation.cif.gz emd_15848_validation.cif.gz | 22.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15848 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15848 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15848 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15848 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b4hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15848.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15848.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15848_msk_1.map emd_15848_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_15848_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15848_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15848_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : IstA transposase cleaved donor complex
| 全体 | 名称: IstA transposase cleaved donor complex |
|---|---|
| 要素 |
|
-超分子 #1: IstA transposase cleaved donor complex
| 超分子 | 名称: IstA transposase cleaved donor complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Complex of IstA transposase bound to two right IS5376 transposon ends |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 225 KDa |
-分子 #1: Putative transposase for insertion sequence element IS5376
| 分子 | 名称: Putative transposase for insertion sequence element IS5376 タイプ: protein_or_peptide / ID: 1 詳細: The residues annotated as cloning artefact are the following five flanking bases of the X67861 gene コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 47.687723 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MITRGEFFMI KEMYERGMSI SDIARELGID RKTVRKYIHS PNPPSKSKRK QRKSKLDPFK PYLQKRMLED GVFNSEKLFF EIRQQGYTG GKTILKDYMK PFRETAKKKY TVRYETLPGE QMQVDWKEVG EVVIEGKKVK LSLFVATLGY SRMKYAVFTT S QDQEHLME ...文字列: MITRGEFFMI KEMYERGMSI SDIARELGID RKTVRKYIHS PNPPSKSKRK QRKSKLDPFK PYLQKRMLED GVFNSEKLFF EIRQQGYTG GKTILKDYMK PFRETAKKKY TVRYETLPGE QMQVDWKEVG EVVIEGKKVK LSLFVATLGY SRMKYAVFTT S QDQEHLME CLIQSFKYFG GVPKKVLFDN MKTVTDGREQ GVVKWNQRFS EFASYYGFIP KVCRPYRAQT KGKVERAIQY IM DHFYVGT AFESIEELNF LLHRWLDQVA NRKPNATTGI SPQERWAEES LKPLPLKDYD TSYLSYRKVH WDGSFSYKGE QWL LSAEYA GKEILVKERL NGDIRLYFRG EEISHVDQQK KVISFAEKIK KKQTEMAATI SPVSVEVDTR PLSVYDAFLR GESS ENLYF Q UniProtKB: Putative transposase for insertion sequence element IS5376 |
-分子 #2: DNA (57-MER) / right IS21 transposon end (insertion sequence IS5376)
| 分子 | 名称: DNA (57-MER) / right IS21 transposon end (insertion sequence IS5376) タイプ: dna / ID: 2 / 詳細: GB X67861 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 18.384814 KDa |
| 配列 | 文字列: (DA)(DT)(DT)(DC)(DA)(DT)(DG)(DT)(DC)(DA) (DA)(DG)(DG)(DC)(DC)(DG)(DA)(DT)(DT)(DA) (DT)(DT)(DT)(DT)(DT)(DT)(DC)(DC)(DC) (DC)(DA)(DA)(DA)(DA)(DT)(DC)(DG)(DC)(DC) (DG) (DG)(DT)(DT)(DT)(DA) ...文字列: (DA)(DT)(DT)(DC)(DA)(DT)(DG)(DT)(DC)(DA) (DA)(DG)(DG)(DC)(DC)(DG)(DA)(DT)(DT)(DA) (DT)(DT)(DT)(DT)(DT)(DT)(DC)(DC)(DC) (DC)(DA)(DA)(DA)(DA)(DT)(DC)(DG)(DC)(DC) (DG) (DG)(DT)(DT)(DT)(DA)(DA)(DA)(DA) (DT)(DT)(DC)(DC)(DC)(DC)(DA)(DG)(DA)(DA) (DG)(DG) |
-分子 #3: DNA (55-MER) / right IS21 transposon end (insertion sequence IS5376)
| 分子 | 名称: DNA (55-MER) / right IS21 transposon end (insertion sequence IS5376) タイプ: dna / ID: 3 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 17.029939 KDa |
| 配列 | 文字列: (DC)(DC)(DT)(DT)(DC)(DT)(DG)(DG)(DG)(DG) (DA)(DA)(DT)(DT)(DT)(DT)(DA)(DA)(DA)(DC) (DC)(DG)(DG)(DC)(DG)(DA)(DT)(DT)(DT) (DT)(DG)(DG)(DG)(DG)(DA)(DA)(DA)(DA)(DA) (DA) (DT)(DA)(DA)(DT)(DC) ...文字列: (DC)(DC)(DT)(DT)(DC)(DT)(DG)(DG)(DG)(DG) (DA)(DA)(DT)(DT)(DT)(DT)(DA)(DA)(DA)(DC) (DC)(DG)(DG)(DC)(DG)(DA)(DT)(DT)(DT) (DT)(DG)(DG)(DG)(DG)(DA)(DA)(DA)(DA)(DA) (DA) (DT)(DA)(DA)(DT)(DC)(DG)(DG)(DC) (DC)(DT)(DT)(DG)(DA)(DC)(DA) GENBANK: GENBANK: X67861.1 |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.119 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 25 mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 3.36 sec. / 平均電子線量: 59.7 e/Å2 / 詳細: Single shot per hole |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 115.818 |
|---|---|
| 得られたモデル |  PDB-8b4h: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)