+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | native Coxsackievirus A9 | |||||||||
 マップデータ マップデータ | Post-processed map of Coxsackievirus A9 in a native state (containing genomic RNA) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | icosahedral symmetry / intact virion / picornavirus / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / nucleoside-triphosphate phosphatase / channel activity ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / DNA replication / RNA helicase activity / induction by virus of host autophagy / RNA-directed RNA polymerase / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / virus-mediated perturbation of host defense response / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / ATP binding / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) / Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) /  Coxsackievirus A9 (コクサッキーウイルス) Coxsackievirus A9 (コクサッキーウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Domanska A / Plavec Z / Ruokolainen V / Marjomaki VS / Butcher SJ | |||||||||
| 資金援助 |  フィンランド, 1件 フィンランド, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2022 ジャーナル: J Virol / 年: 2022タイトル: Structural Studies Reveal that Endosomal Cations Promote Formation of Infectious Coxsackievirus A9 A-Particles, Facilitating RNA and VP4 Release. 著者: Aušra Domanska / Zlatka Plavec / Visa Ruokolainen / Benita Löflund / Varpu Marjomäki / Sarah J Butcher /  要旨: Coxsackievirus A9 (CVA9), an enterovirus, is a common cause of pediatric aseptic meningitis and neonatal sepsis. During cell entry, enterovirus capsids undergo conformational changes leading to ...Coxsackievirus A9 (CVA9), an enterovirus, is a common cause of pediatric aseptic meningitis and neonatal sepsis. During cell entry, enterovirus capsids undergo conformational changes leading to expansion, formation of large pores, externalization of VP1 N termini, and loss of the lipid factor from VP1. Factors such as receptor binding, heat, and acidic pH can trigger capsid expansion in some enteroviruses. Here, we show that fatty acid-free bovine serum albumin or neutral endosomal ionic conditions can independently prime CVA9 for expansion and genome release. Our results showed that CVA9 treatment with albumin or endosomal ions generated a heterogeneous population of virions, which could be physically separated by asymmetric flow field flow fractionation and computationally by cryo-electron microscopy (cryo-EM) and image processing. We report cryo-EM structures of CVA9 A-particles obtained by albumin or endosomal ion treatment and a control nonexpanded virion to 3.5, 3.3, and 2.9 Å resolution, respectively. Whereas albumin promoted stable expanded virions, the endosomal ionic concentrations induced unstable CVA9 virions which easily disintegrated, losing their genome. Loss of most of the VP4 molecules and exposure of negatively charged amino acid residues in the capsid's interior after expansion created a repulsive viral RNA-capsid interface, aiding genome release. Coxsackievirus A9 (CVA9) is a common cause of meningitis and neonatal sepsis. The triggers and mode of action of RNA release into the cell unusually do not require receptor interaction. Rather, a slow process in the endosome, independent of low pH, is required. Here, we show by biophysical separation, cryogenic electron microscopy, and image reconstruction that albumin and buffers mimicking the endosomal ion composition can separately and together expand and prime CVA9 for uncoating. Furthermore, we show in these expanded particles that VP4 is present at only ~10% of the occupancy found in the virion, VP1 is externalized, and the genome is repelled by the negatively charged, repulsive inner surface of the capsid that occurs due to the expansion. Thus, we can now link observations from cell biology of infection with the physical processes that occur in the capsid to promote genome uncoating. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15634.map.gz emd_15634.map.gz | 73 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15634-v30.xml emd-15634-v30.xml emd-15634.xml emd-15634.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15634.png emd_15634.png | 199.6 KB | ||
| Filedesc metadata |  emd-15634.cif.gz emd-15634.cif.gz | 6.6 KB | ||
| その他 |  emd_15634_half_map_1.map.gz emd_15634_half_map_1.map.gz emd_15634_half_map_2.map.gz emd_15634_half_map_2.map.gz | 191.7 MB 191.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15634 http://ftp.pdbj.org/pub/emdb/structures/EMD-15634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15634 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15634_validation.pdf.gz emd_15634_validation.pdf.gz | 986.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15634_full_validation.pdf.gz emd_15634_full_validation.pdf.gz | 986.5 KB | 表示 | |
| XML形式データ |  emd_15634_validation.xml.gz emd_15634_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_15634_validation.cif.gz emd_15634_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15634 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15634 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15634 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15634 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15634.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15634.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map of Coxsackievirus A9 in a native state (containing genomic RNA) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.97 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map1 of Coxsackievirus A9 in a native...
| ファイル | emd_15634_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
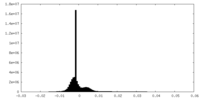
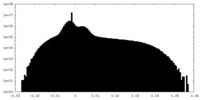
| 注釈 | Half map1 of Coxsackievirus A9 in a native state (containing genomic RNA) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map2 of Coxsackievirus A9 in a native...
| ファイル | emd_15634_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
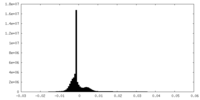
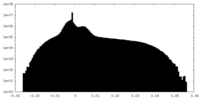
| 注釈 | Half map2 of Coxsackievirus A9 in a native state (containing genomic RNA) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Coxsackievirus A9
| 全体 | 名称:  Coxsackievirus A9 (コクサッキーウイルス) Coxsackievirus A9 (コクサッキーウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Coxsackievirus A9
| 超分子 | 名称: Coxsackievirus A9 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #2-#4, #1 / NCBI-ID: 12067 / 生物種: Coxsackievirus A9 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: icosahedral / 直径: 300.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 1 / 詳細: Viral protein 4, VP4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス)株: Griggs |
| 分子量 | 理論値: 7.352039 KDa |
| 配列 | 文字列: GAQVSTQKTG AHETSLSAAG NSIIHYTNIN YYKDAASNSA NRQDFTQDPS KFTEPVKDVM IKSLPALN UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 2 / 詳細: Viral protein 1, VP1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス)株: Griggs / 器官: kidney |
| 分子量 | 理論値: 33.86902 KDa |
| 配列 | 文字列: GDVEEAIERA VVHVADTMRS GPSNSASVPA LTAVETGHTS QVTPSDTMQT RHVKNYHSRS ESTVENFLGR SACVYMEEYK TTDNDVNKK FVAWPINTKQ MVQMRRKLEM FTYLRFDMEV TFVITSRQDP GTTLAQDMPV LTHQIMYVPP GGPIPAKVDD Y AWQTSTNP ...文字列: GDVEEAIERA VVHVADTMRS GPSNSASVPA LTAVETGHTS QVTPSDTMQT RHVKNYHSRS ESTVENFLGR SACVYMEEYK TTDNDVNKK FVAWPINTKQ MVQMRRKLEM FTYLRFDMEV TFVITSRQDP GTTLAQDMPV LTHQIMYVPP GGPIPAKVDD Y AWQTSTNP SIFWTEGNAP ARMSIPFISI GNAYSNFYDG WSNFDQRGSY GYNTLNNLGH IYVRHVSGSS PHPITSTIRV YF KPKHTRA WVPRPPRLCQ YKKAFSVDFT PTPITDTRKD INTVTTVAQS RRRGDMSTLN TH UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 3 / 詳細: Viral protein 2, VP2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス)株: Griggs |
| 分子量 | 理論値: 28.885518 KDa |
| 配列 | 文字列: SPTVEECGYS DRVRSITLGN STITTQECAN VVVGYGRWPT YLRDDEATAE DQPTQPDVAT CRFYTLDSIK WEKGSVGWWW KFPEALSDM GLFGQNMQYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGGAVVGQ AFSATAMANG DKAYEFTSAT Q SDQTKVQT ...文字列: SPTVEECGYS DRVRSITLGN STITTQECAN VVVGYGRWPT YLRDDEATAE DQPTQPDVAT CRFYTLDSIK WEKGSVGWWW KFPEALSDM GLFGQNMQYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGGAVVGQ AFSATAMANG DKAYEFTSAT Q SDQTKVQT AIHNAGMGVG VGNLTIYPHQ WINLRTNNSA TIVMPYINSV PMDNMFRHYN FTLMVIPFVK LDYADTASTY VP ITVTVAP MCAEYNGLRL AQAQ UniProtKB: Genome polyprotein |
-分子 #4: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 4 / 詳細: Viral protein 3, VP3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス) Human coxsackievirus A9 (strain Griggs) (コクサッキーウイルス)株: Griggs |
| 分子量 | 理論値: 26.335072 KDa |
| 配列 | 文字列: GLPTMNTPGS TQFLTSDDFQ SPCALPQFDV TPSMNIPGEV KNLMEIAEVD SVVPVNNVQD TTDQMEMFRI PVTINAPLQQ QVFGLRLQP GLDSVFKHTL LGEILNYYAH WSGSMKLTFV FCGSAMATGK FLIAYSPPGA NPPKTRKDAM LGTHIIWDIG L QSSCVLCV ...文字列: GLPTMNTPGS TQFLTSDDFQ SPCALPQFDV TPSMNIPGEV KNLMEIAEVD SVVPVNNVQD TTDQMEMFRI PVTINAPLQQ QVFGLRLQP GLDSVFKHTL LGEILNYYAH WSGSMKLTFV FCGSAMATGK FLIAYSPPGA NPPKTRKDAM LGTHIIWDIG L QSSCVLCV PWISQTHYRL VQQDEYTSAG YVTCWYQTGM IVPPGTPNSS SIMCFASACN DFSVRMLRDT PFISQDNKLQ UniProtKB: Genome polyprotein |
-分子 #5: MYRISTIC ACID
| 分子 | 名称: MYRISTIC ACID / タイプ: ligand / ID: 5 / コピー数: 1 / 式: MYR |
|---|---|
| 分子量 | 理論値: 228.371 Da |
| Chemical component information |  ChemComp-MYR: |
-分子 #6: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 6 / コピー数: 1 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.25 / 詳細: PBS containing 2mM MgCl2, pH 7.2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
| 詳細 | this sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 150000 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)