+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 305 A SynPspA rod after incubation with ATP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleotide binding / Helical assembly / ESCRT-III fold / Membrane remodeling / LIPID BINDING PROTEIN | |||||||||
| 機能・相同性 | PspA/IM30 / PspA/IM30 family / Chloroplast membrane-associated 30 kD protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
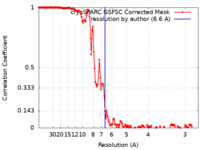
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 6.6 Å | |||||||||
 データ登録者 データ登録者 | Junglas B / Hudina E / Schoennenbeck P / Ritter I / Santiago-Schuebel B / Huesgen P / Sachse C | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural plasticity of bacterial ESCRT-III protein PspA in higher-order assemblies. 著者: Benedikt Junglas / Esther Hudina / Philipp Schönnenbeck / Ilona Ritter / Anja Heddier / Beatrix Santiago-Schübel / Pitter F Huesgen / Dirk Schneider / Carsten Sachse /  要旨: Eukaryotic members of the endosome sorting complex required for transport-III (ESCRT-III) family have been shown to form diverse higher-order assemblies. The bacterial phage shock protein A (PspA) ...Eukaryotic members of the endosome sorting complex required for transport-III (ESCRT-III) family have been shown to form diverse higher-order assemblies. The bacterial phage shock protein A (PspA) has been identified as a member of the ESCRT-III superfamily, and PspA homo-oligomerizes to form rod-shaped assemblies. As observed for eukaryotic ESCRT-III, PspA forms tubular assemblies of varying diameters. Using electron cryo-electron microscopy, we determined 61 Synechocystis PspA structures and observed in molecular detail how the structural plasticity of PspA rods is mediated by conformational changes at three hinge regions in the monomer and by the fixed and changing molecular contacts between protomers. Moreover, we reduced and increased the structural plasticity of PspA rods by removing the loop connecting helices α3/α4 and the addition of nucleotides, respectively. Based on our analysis of PspA-mediated membrane remodeling, we suggest that the observed mode of structural plasticity is a prerequisite for the biological function of ESCRT-III members. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15496.map.gz emd_15496.map.gz | 8.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15496-v30.xml emd-15496-v30.xml emd-15496.xml emd-15496.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15496_fsc.xml emd_15496_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15496.png emd_15496.png | 49.4 KB | ||
| マスクデータ |  emd_15496_msk_1.map emd_15496_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15496.cif.gz emd-15496.cif.gz | 5.4 KB | ||
| その他 |  emd_15496_additional_1.map.gz emd_15496_additional_1.map.gz emd_15496_half_map_1.map.gz emd_15496_half_map_1.map.gz emd_15496_half_map_2.map.gz emd_15496_half_map_2.map.gz | 36.2 MB 226.1 MB 226.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15496 http://ftp.pdbj.org/pub/emdb/structures/EMD-15496 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15496 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15496 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15496_validation.pdf.gz emd_15496_validation.pdf.gz | 1005.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15496_full_validation.pdf.gz emd_15496_full_validation.pdf.gz | 1005.1 KB | 表示 | |
| XML形式データ |  emd_15496_validation.xml.gz emd_15496_validation.xml.gz | 22.2 KB | 表示 | |
| CIF形式データ |  emd_15496_validation.cif.gz emd_15496_validation.cif.gz | 29 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15496 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15496 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15496 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15496 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8akxMC  8akqC  8akrC  8aksC  8aktC  8akuC  8akvC  8akwC  8akyC  8akzC  8al0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15496.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15496.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.362 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15496_msk_1.map emd_15496_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local filtered map
| ファイル | emd_15496_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15496_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15496_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Phage shock protein A (PspA)
| 全体 | 名称: Phage shock protein A (PspA) |
|---|---|
| 要素 |
|
-超分子 #1: Phage shock protein A (PspA)
| 超分子 | 名称: Phage shock protein A (PspA) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 162.2 kDa/nm |
-分子 #1: Chloroplast membrane-associated 30 kD protein
| 分子 | 名称: Chloroplast membrane-associated 30 kD protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 60 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.097758 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSSAALEVLF QGPMELFNRV GRVLKSQLTH WQQQQEAPED LLERLLGEME LELIELRRAL AQTIATFKST ERQRDAQQL IAQRWYEKAQ AALDRGNEQL AREALGQRQS YQSHTEALGK SLGEQRALVE QVRGQLQKLE RKYLELKSQK N LYLARLKS ...文字列: MGSSHHHHHH SSSAALEVLF QGPMELFNRV GRVLKSQLTH WQQQQEAPED LLERLLGEME LELIELRRAL AQTIATFKST ERQRDAQQL IAQRWYEKAQ AALDRGNEQL AREALGQRQS YQSHTEALGK SLGEQRALVE QVRGQLQKLE RKYLELKSQK N LYLARLKS AIAAQKIEEI AGNLDNASAS SLFERIETKI LELEAERELL NPPPSPLDKK FEQWEEQQAV EATLAAMKAR RS LPPPSS UniProtKB: Chloroplast membrane-associated 30 kD protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)