+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Complex of RecO-RecR-DNA from Thermus thermophilus. | |||||||||
 マップデータ マップデータ | map sharpened locally with Local Filter | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Thermus thermophilus HB8 (バクテリア) / synthetic construct (人工物) Thermus thermophilus HB8 (バクテリア) / synthetic construct (人工物) | |||||||||
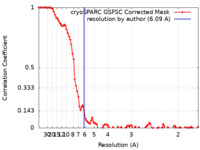
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.09 Å | |||||||||
 データ登録者 データ登録者 | Nirwal S / Czarnocki-Cieciura M / Chaudhary A / Zajko W / Skowronek K / Chamera S / Figiel M / Nowotny M | |||||||||
| 資金援助 |  ポーランド, 1件 ポーランド, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Mechanism of RecF-RecO-RecR cooperation in bacterial homologous recombination. 著者: Shivlee Nirwal / Mariusz Czarnocki-Cieciura / Anuradha Chaudhary / Weronika Zajko / Krzysztof Skowronek / Sebastian Chamera / Małgorzata Figiel / Marcin Nowotny /  要旨: In bacteria, one type of homologous-recombination-based DNA-repair pathway involves RecFOR proteins that bind at the junction between single-stranded (ss) and double-stranded (ds) DNA. They ...In bacteria, one type of homologous-recombination-based DNA-repair pathway involves RecFOR proteins that bind at the junction between single-stranded (ss) and double-stranded (ds) DNA. They facilitate the replacement of SSB protein, which initially covers ssDNA, with RecA, which mediates the search for homologous sequences. However, the molecular mechanism of RecFOR cooperation remains largely unknown. We used Thermus thermophilus proteins to study this system. Here, we present a cryo-electron microscopy structure of the RecF-dsDNA complex, and another reconstruction that shows how RecF interacts with two different regions of the tetrameric RecR ring. Lower-resolution reconstructions of the RecR-RecO subcomplex and the RecFOR-DNA assembly explain how RecO is positioned to interact with ssDNA and SSB, which is proposed to lock the complex on a ssDNA-dsDNA junction. Our results integrate the biochemical data available for the RecFOR system and provide a framework for its complete understanding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15308.map.gz emd_15308.map.gz | 3.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15308-v30.xml emd-15308-v30.xml emd-15308.xml emd-15308.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15308_fsc.xml emd_15308_fsc.xml | 9.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15308.png emd_15308.png | 75.4 KB | ||
| マスクデータ |  emd_15308_msk_1.map emd_15308_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15308_additional_1.map.gz emd_15308_additional_1.map.gz emd_15308_half_map_1.map.gz emd_15308_half_map_1.map.gz emd_15308_half_map_2.map.gz emd_15308_half_map_2.map.gz | 40.2 MB 77.6 MB 77.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15308 http://ftp.pdbj.org/pub/emdb/structures/EMD-15308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15308 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15308_validation.pdf.gz emd_15308_validation.pdf.gz | 680.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15308_full_validation.pdf.gz emd_15308_full_validation.pdf.gz | 679.7 KB | 表示 | |
| XML形式データ |  emd_15308_validation.xml.gz emd_15308_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_15308_validation.cif.gz emd_15308_validation.cif.gz | 22.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15308 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15308 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15308.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15308.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map sharpened locally with Local Filter | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15308_msk_1.map emd_15308_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
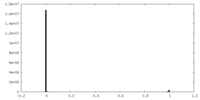
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: raw map
| ファイル | emd_15308_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | raw map | ||||||||||||
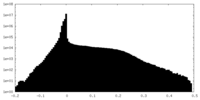
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_15308_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
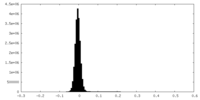
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_15308_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
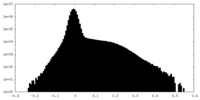
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of RecO-RecR-DNA from Thermus thermophilus.
| 全体 | 名称: Complex of RecO-RecR-DNA from Thermus thermophilus. |
|---|---|
| 要素 |
|
-超分子 #1: Complex of RecO-RecR-DNA from Thermus thermophilus.
| 超分子 | 名称: Complex of RecO-RecR-DNA from Thermus thermophilus. / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus HB8 (バクテリア) Thermus thermophilus HB8 (バクテリア) |
| 分子量 | 理論値: 130 KDa |
-分子 #1: Recombination protein RecR
| 分子 | 名称: Recombination protein RecR / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus HB8 (バクテリア) / 株: ATCC 27634 / DSM 579 / HB8 Thermus thermophilus HB8 (バクテリア) / 株: ATCC 27634 / DSM 579 / HB8 |
| 分子量 | 理論値: 21.323467 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SMRYPESLLK LTRALSRLPG IGPKTAQRLA LHLAFHKEEA EALAEALEGI KRVRACRECG NLAEGELCPI CQDEDRDRSL LAVVESVAD LYALERSGEF RGLYHVLGGA LNPLEGIGPK ELNLEGLFRR LEGVEEVVLA TSMTVEGEAT ALYLAEELKK R GVRVTRPA ...文字列: SMRYPESLLK LTRALSRLPG IGPKTAQRLA LHLAFHKEEA EALAEALEGI KRVRACRECG NLAEGELCPI CQDEDRDRSL LAVVESVAD LYALERSGEF RGLYHVLGGA LNPLEGIGPK ELNLEGLFRR LEGVEEVVLA TSMTVEGEAT ALYLAEELKK R GVRVTRPA YGLPVGGSLE YADEVTLGRA LEGRRPV |
-分子 #2: DNA repair protein RecO
| 分子 | 名称: DNA repair protein RecO / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus HB8 (バクテリア) / 株: ATCC 27634 / DSM 579 / HB8 Thermus thermophilus HB8 (バクテリア) / 株: ATCC 27634 / DSM 579 / HB8 |
| 分子量 | 理論値: 24.928121 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SMAPAYTGKV ERYRLEEGIV VGRKPLPQGD LLLRLVTPRG SLEAVVRKGQ RPTGRTGRLS LFHHVRFQLY AKGEGLPTLT QAELLGRLH GLEAPRRFLL AAFLAELAYR LASPEAAPRI YPLLVSGLRG IAKHEDPLLP LVWAGWRVAK AGGIGPNLEG E GLRLKRGR ...文字列: SMAPAYTGKV ERYRLEEGIV VGRKPLPQGD LLLRLVTPRG SLEAVVRKGQ RPTGRTGRLS LFHHVRFQLY AKGEGLPTLT QAELLGRLH GLEAPRRFLL AAFLAELAYR LASPEAAPRI YPLLVSGLRG IAKHEDPLLP LVWAGWRVAK AGGIGPNLEG E GLRLKRGR LGEEGVYLGR EGVEALKATL RLPGAQALPH LEGAPLNRLF LALKAHAEEA LGPLRSAEAI GV |
-分子 #3: Oligo1
| 分子 | 名称: Oligo1 / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.661912 KDa |
| 配列 | 文字列: (DG)(DG)(DC)(DC)(DA)(DG)(DA)(DT)(DC)(DT) (DG)(DC)(DC)(DG)(DC)(DG)(DG)(DA)(DT)(DC) (DC)(DG)(DC)(DG)(DC) |
-分子 #4: Oligo2
| 分子 | 名称: Oligo2 / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.395925 KDa |
| 配列 | 文字列: (DG)(DC)(DG)(DC)(DG)(DG)(DA)(DT)(DC)(DC) (DG)(DC)(DG)(DG)(DC)(DA)(DG)(DA)(DT)(DC) (DT)(DG)(DG)(DC)(DC)(DT)(DG)(DA)(DT) (DT)(DG)(DC)(DG)(DG)(DT)(DA)(DC)(DA)(DG) (DA) |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | Sample fixed with 0.05% glutaraldehyde and concentrated prior to vitrification; exact concentration cannot be estimated accurately. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 7217 / 平均電子線量: 41.71 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8ab0: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X