+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Solution BcsD structure | |||||||||
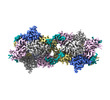
 マップデータ マップデータ | BcsD octamer in solution : Unsharpened cryo-EM map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Cellulose synthase operon protein D, bacterial / Cellulose synthase subunit D superfamily / Cellulose synthase subunit D / cellulose biosynthetic process / Cellulose biosynthesis protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Komagataeibacter hansenii ATCC 23769 (バクテリア) Komagataeibacter hansenii ATCC 23769 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Krasteva PV / Abidi W / Decossas M | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Bacterial crystalline cellulose secretion via a supramolecular BcsHD scaffold. 著者: Wiem Abidi / Marion Decossas / Lucía Torres-Sánchez / Lucie Puygrenier / Sylvie Létoffé / Jean-Marc Ghigo / Petya V Krasteva /  要旨: Cellulose, the most abundant biopolymer on Earth, is not only the predominant constituent of plants but also a key extracellular polysaccharide in the biofilms of many bacterial species. Depending on ...Cellulose, the most abundant biopolymer on Earth, is not only the predominant constituent of plants but also a key extracellular polysaccharide in the biofilms of many bacterial species. Depending on the producers, chemical modifications, and three-dimensional assemblies, bacterial cellulose (BC) can present diverse degrees of crystallinity. Highly ordered, or crystalline, cellulose presents great economical relevance due to its ever-growing number of biotechnological applications. Even if some acetic acid bacteria have long been identified as BC superproducers, the molecular mechanisms determining the secretion of crystalline versus amorphous cellulose remain largely unknown. Here, we present structural and mechanistic insights into the role of the accessory subunits BcsH (CcpAx) and BcsD (CesD) that determine crystalline BC secretion in the lineage. We show that oligomeric BcsH drives the assembly of BcsD into a supramolecular cytoskeletal scaffold that likely stabilizes the cellulose-extruding synthase nanoarrays through an unexpected inside-out mechanism for secretion system assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15041.map.gz emd_15041.map.gz | 61.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15041-v30.xml emd-15041-v30.xml emd-15041.xml emd-15041.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15041_fsc.xml emd_15041_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15041.png emd_15041.png | 48.2 KB | ||
| マスクデータ |  emd_15041_msk_1.map emd_15041_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15041_additional_1.map.gz emd_15041_additional_1.map.gz emd_15041_half_map_1.map.gz emd_15041_half_map_1.map.gz emd_15041_half_map_2.map.gz emd_15041_half_map_2.map.gz | 117.6 MB 115.6 MB 115.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15041 http://ftp.pdbj.org/pub/emdb/structures/EMD-15041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15041 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15041_validation.pdf.gz emd_15041_validation.pdf.gz | 571.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15041_full_validation.pdf.gz emd_15041_full_validation.pdf.gz | 571 KB | 表示 | |
| XML形式データ |  emd_15041_validation.xml.gz emd_15041_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_15041_validation.cif.gz emd_15041_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15041 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15041 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15041.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15041.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BcsD octamer in solution : Unsharpened cryo-EM map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15041_msk_1.map emd_15041_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
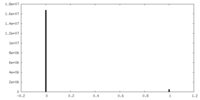
| 密度ヒストグラム |
-追加マップ: BcsD octamer in solution : Autosharpened cryo-EM map (cryoSPARC)
| ファイル | emd_15041_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BcsD octamer in solution : Autosharpened cryo-EM map (cryoSPARC) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
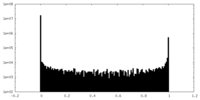
| 密度ヒストグラム |
-ハーフマップ: BcsD octamer in solution : half map A
| ファイル | emd_15041_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BcsD octamer in solution : half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
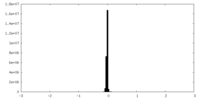
| 密度ヒストグラム |
-ハーフマップ: BcsD octamer in solution : half map B
| ファイル | emd_15041_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BcsD octamer in solution : half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
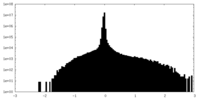
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BcsD from G. hansenii
| 全体 | 名称: BcsD from G. hansenii |
|---|---|
| 要素 |
|
-超分子 #1: BcsD from G. hansenii
| 超分子 | 名称: BcsD from G. hansenii / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 詳細: Octameric BcsD in solution. Expressed recombinantly in E. coli and purified via a cleavable N-terminal hexahistidine tag. |
|---|---|
| 由来(天然) | 生物種:  Komagataeibacter hansenii ATCC 23769 (バクテリア) Komagataeibacter hansenii ATCC 23769 (バクテリア)株: ATCC 23769 |
-分子 #1: Cellulose biosynthesis protein
| 分子 | 名称: Cellulose biosynthesis protein / タイプ: protein_or_peptide / ID: 1 詳細: The HRV3c-cleavable hexahistidine tag was cleaved during the purification process. Sample protein sequence starts with PMGSTIFEK... コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Komagataeibacter hansenii ATCC 23769 (バクテリア) Komagataeibacter hansenii ATCC 23769 (バクテリア) |
| 分子量 | 理論値: 20.596332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTLEV LFQGPMGSTI FEKKPDFTLF LQTLSWEIDD QVGIEVRNEL LREVGRGMGT RIMPPPCQTV DKLQIELNA LLALIGWGTV TLELLSEDQS LRIVHENLPQ VGSAGEPSGT WLAPVLEGLY GRWVTSQAGA FGDYVVTRDV D AEDLNAVP RQTIIMYMRV RSSAT |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 24 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20 mM HEPES pH 8.0, 100 mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.59 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.75 µm / 最小 デフォーカス(公称値): 0.48 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X