+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of "CT pyr" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Tetramer / carboxylase / biotin / inhibitor / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pyruvate carboxylase / pyruvate carboxylase activity / pyruvate metabolic process / gluconeogenesis / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) | |||||||||
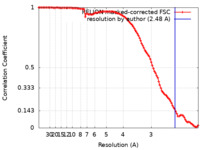
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.48 Å | |||||||||
 データ登録者 データ登録者 | Lopez-Alonso JP / Lazaro M / Gil D / Choi PH / Tong L / Valle M | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: CryoEM structural exploration of catalytically active enzyme pyruvate carboxylase. 著者: Jorge Pedro López-Alonso / Melisa Lázaro / David Gil-Cartón / Philip H Choi / Alexandra Dodu / Liang Tong / Mikel Valle /   要旨: Pyruvate carboxylase (PC) is a tetrameric enzyme that contains two active sites per subunit that catalyze two consecutive reactions. A mobile domain with an attached prosthetic biotin links both ...Pyruvate carboxylase (PC) is a tetrameric enzyme that contains two active sites per subunit that catalyze two consecutive reactions. A mobile domain with an attached prosthetic biotin links both reactions, an initial biotin carboxylation and the subsequent carboxyl transfer to pyruvate substrate to produce oxaloacetate. Reaction sites are at long distance, and there are several co-factors that play as allosteric regulators. Here, using cryoEM we explore the structure of active PC tetramers focusing on active sites and on the conformational space of the oligomers. The results capture the mobile domain at both active sites and expose catalytic steps of both reactions at high resolution, allowing the identification of substrates and products. The analysis of catalytically active PC tetramers reveals the role of certain motions during enzyme functioning, and the structural changes in the presence of additional cofactors expose the mechanism for allosteric regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15032.map.gz emd_15032.map.gz | 940.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15032-v30.xml emd-15032-v30.xml emd-15032.xml emd-15032.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15032_fsc.xml emd_15032_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15032.png emd_15032.png | 97.5 KB | ||
| マスクデータ |  emd_15032_msk_1.map emd_15032_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15032.cif.gz emd-15032.cif.gz | 7 KB | ||
| その他 |  emd_15032_half_map_1.map.gz emd_15032_half_map_1.map.gz emd_15032_half_map_2.map.gz emd_15032_half_map_2.map.gz | 80.9 MB 80.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15032 http://ftp.pdbj.org/pub/emdb/structures/EMD-15032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15032 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15032_validation.pdf.gz emd_15032_validation.pdf.gz | 628.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15032_full_validation.pdf.gz emd_15032_full_validation.pdf.gz | 628.5 KB | 表示 | |
| XML形式データ |  emd_15032_validation.xml.gz emd_15032_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_15032_validation.cif.gz emd_15032_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15032 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15032 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zz2MC  7zyyC  7zyzC  7zz0C  7zz1C  7zz3C  7zz4C  7zz5C  7zz6C  7zz8C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15032.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15032.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
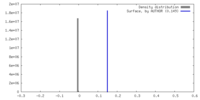
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15032_msk_1.map emd_15032_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
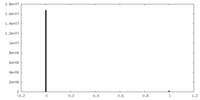
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15032_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
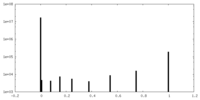
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15032_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
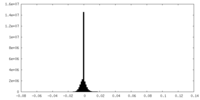
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pyruvate carboxylase with acetyl coenzyme A
| 全体 | 名称: Pyruvate carboxylase with acetyl coenzyme A |
|---|---|
| 要素 |
|
-超分子 #1: Pyruvate carboxylase with acetyl coenzyme A
| 超分子 | 名称: Pyruvate carboxylase with acetyl coenzyme A / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
-分子 #1: Pyruvate carboxylase
| 分子 | 名称: Pyruvate carboxylase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: pyruvate carboxylase |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
| 分子量 | 理論値: 127.349578 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VPRGSHMKKL LVANRGEIAV RVFRACNELG LSTVAVYARE DEYSVHRFKA DESYLIGQGK KPIDAYLDID DIIRVALESG ADAIHPGYG LLSENLEFAT KVRAAGLVFV GPELHHLDIF GDKIKAKAAA DEAKVPGIPG TNGAVDIDGA LEFAKTYGYP V MIKAALGG ...文字列: VPRGSHMKKL LVANRGEIAV RVFRACNELG LSTVAVYARE DEYSVHRFKA DESYLIGQGK KPIDAYLDID DIIRVALESG ADAIHPGYG LLSENLEFAT KVRAAGLVFV GPELHHLDIF GDKIKAKAAA DEAKVPGIPG TNGAVDIDGA LEFAKTYGYP V MIKAALGG GGRGMRVARN DAEMHDGYAR AKSEAIGAFG SGEIYVEKYI ENPKHIEVQI LGDRHGNIIH LHERDCSVQR RN QKVIEIA PAVGLSPDFR NEICEAAVKL CKNVGYVNAG TVEFLVKDDK FYFIEVNPRV QVEHTITELI TGVDIVQAQI LIA QGKDLH REIGLPAQSE IPLLGSAIQC RITTEDPQNG FLPDTGKIDT YRSPGGFGIR LDVGNAYAGY EVTPYFDSLL VKVC TFANE FSDSVRKMDR VLHEFRIRGV KTNIPFLINV IANENFTSGQ ATTTFIDNTP SLFNFPRLRD RGTKTLHYLS MITVN GFPG IENTEKRHFE EPRQPLLNLE KKKTAKNILD EQGADAVVDY VKNTKEVLLT DTTLRDAHQS LLATRLRLQD MKGIAQ AID QGLPELFSAE MWGGATFDVA YRFLNESPWY RLRKLRKLMP NTMFQMLFRG SNAVGYQNYP DNVIEEFIRV AAHEGID VF RIFDSLNWLP QMEKSIQAVR DNGKIAEATI CYTGDILDPS RPKYNIQYYK DLAKELEATG AHILAV(KCX)DMA GLLK PQAAY RLISELKDTV DLPIHLHTHD TSGNGIITYS GATQAGVDII DVATASLAGG TSQPSMQSIY YALEHGPRHA SINVK NAEQ IDHYWEDVRK YYAPFEAGIT SPQTEVYMHE MPGGQYTNLK SQAAAVGLGH RFDEIKQMYR KVNMMFGDII KVTPSS KVV GDMALFMIQN DLTEEDVYAR GNELNFPESV VSFFRGDLGQ PVGGFPEKLQ KIIVKDKAVI TDRPGLHAEK VDFETVK AD LEQKIGYEPG DHEVISYIMY PQVFLDYQKM QREFGAVTLL DTPTFLHGMR LNEKIEVQIE KGKTLSIRLD EIGEPDLA G NRVLFFNLNG QRREVVINDQ SVQAQVVAKR KAETGNPNQI GATMPGSVLE ILVKAGDKVQ KGQALMVTEA MKMETTIEA PFDGEIVDLH VVKGEAIQTQ DLLIEIN UniProtKB: Pyruvate carboxylase |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #4: PYRUVIC ACID
| 分子 | 名称: PYRUVIC ACID / タイプ: ligand / ID: 4 / コピー数: 1 / 式: PYR |
|---|---|
| 分子量 | 理論値: 88.062 Da |
| Chemical component information | 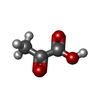 ChemComp-PYR: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 57 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| |||||||||||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 10518 / 平均露光時間: 3.99 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)