+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the human sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody | |||||||||
 マップデータ マップデータ | Nanodisc-reconstituted human NTCP in a complex with NTCP_Fab12 and a nanobody in the presence of substrate. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bile acid:sodium symporter activity / regulation of bile acid secretion / bile acid and bile salt transport / bile acid signaling pathway / Recycling of bile acids and salts / response to nutrient levels / response to organic cyclic compound / response to estrogen / cellular response to xenobiotic stimulus / virus receptor activity ...bile acid:sodium symporter activity / regulation of bile acid secretion / bile acid and bile salt transport / bile acid signaling pathway / Recycling of bile acids and salts / response to nutrient levels / response to organic cyclic compound / response to estrogen / cellular response to xenobiotic stimulus / virus receptor activity / basolateral plasma membrane / response to ethanol / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   | |||||||||
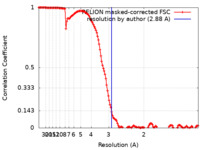
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.88 Å | |||||||||
 データ登録者 データ登録者 | Liu H / Irobalieva RN / Bang-Sorensen R / Nosol K / Mukherjee S / Agrawal P / Stieger B / Kossiakoff AA / Locher KP | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2022 ジャーナル: Cell Res / 年: 2022タイトル: Structure of human NTCP reveals the basis of recognition and sodium-driven transport of bile salts into the liver. 著者: Hongtao Liu / Rossitza N Irobalieva / Rose Bang-Sørensen / Kamil Nosol / Somnath Mukherjee / Parth Agrawal / Bruno Stieger / Anthony A Kossiakoff / Kaspar P Locher /   | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15024.map.gz emd_15024.map.gz | 201.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15024-v30.xml emd-15024-v30.xml emd-15024.xml emd-15024.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15024_fsc.xml emd_15024_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15024.png emd_15024.png | 91.5 KB | ||
| その他 |  emd_15024_half_map_1.map.gz emd_15024_half_map_1.map.gz emd_15024_half_map_2.map.gz emd_15024_half_map_2.map.gz | 171.4 MB 171.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15024 http://ftp.pdbj.org/pub/emdb/structures/EMD-15024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15024 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15024_validation.pdf.gz emd_15024_validation.pdf.gz | 824.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15024_full_validation.pdf.gz emd_15024_full_validation.pdf.gz | 824.2 KB | 表示 | |
| XML形式データ |  emd_15024_validation.xml.gz emd_15024_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_15024_validation.cif.gz emd_15024_validation.cif.gz | 27.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15024 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15024 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15024 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15024 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zyiMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15024.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15024.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nanodisc-reconstituted human NTCP in a complex with NTCP_Fab12 and a nanobody in the presence of substrate. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.66 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_15024_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
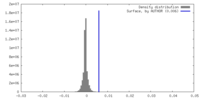
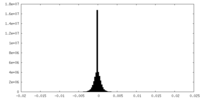
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15024_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
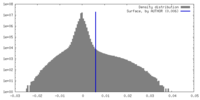
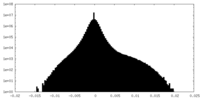
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody
| 全体 | 名称: sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody |
|---|---|
| 要素 |
|
-超分子 #1: sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody
| 超分子 | 名称: sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
| 分子量 | 理論値: 110 KDa |
-分子 #1: Sodium/bile acid cotransporter
| 分子 | 名称: Sodium/bile acid cotransporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 38.149949 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEAHNASAPF NFTLPPNFGK RPTDLALSVI LVFMLFFIML SLGCTMEFSK IKAHLWKPKG LAIALVAQYG IMPLTAFVLG KVFRLKNIE ALAILVCGCS PGGNLSNVFS LAMKGDMNLS IVMTTCSTFC ALGMMPLLLY IYSRGIYDGD LKDKVPYKGI V ISLVLVLI ...文字列: MEAHNASAPF NFTLPPNFGK RPTDLALSVI LVFMLFFIML SLGCTMEFSK IKAHLWKPKG LAIALVAQYG IMPLTAFVLG KVFRLKNIE ALAILVCGCS PGGNLSNVFS LAMKGDMNLS IVMTTCSTFC ALGMMPLLLY IYSRGIYDGD LKDKVPYKGI V ISLVLVLI PCTIGIVLKS KRPQYMRYVI KGGMIIILLC SVAVTVLSAI NVGKSIMFAM TPLLIATSSL MPFIGFLLGY VL SALFCLN GRCRRTVSME TGCQNVQLCS TILNVAFPPE VIGPLFFFPL LYMIFQLGEG LLLIAIFWCY EKFKTPKDKT KMI YTAATT EETIPGALGN GTYKGEDCSP CTA |
-分子 #2: heavy chain of Fab
| 分子 | 名称: heavy chain of Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.582521 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVSYSSIHWV RQAPGKGLEW VASISSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARYMKQQSQM WYQRYWGFDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVSYSSIHWV RQAPGKGLEW VASISSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARYMKQQSQM WYQRYWGFDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKTHT |
-分子 #3: light chain of Fab
| 分子 | 名称: light chain of Fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.417971 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSYWSPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSYWSPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: Nanobody
| 分子 | 名称: Nanobody / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.118386 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VQLQESGGGL VQPGGSLRLS CAASGRTISR YAMSWFRQAP GKEREFVAVA RRSGDGAFYA DSVQGRFTVS RDDAKNTVYL QMNSLKPED TAVYYCAIDS DTFYSGSYDY WGQGTQVTVS S |
-分子 #5: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 5 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #6: GLYCOCHENODEOXYCHOLIC ACID
| 分子 | 名称: GLYCOCHENODEOXYCHOLIC ACID / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CHO |
|---|---|
| 分子量 | 理論値: 449.623 Da |
| Chemical component information | 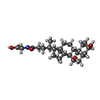 ChemComp-CHO: |
-分子 #7: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 7 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 13208 / 平均露光時間: 2.0 sec. / 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)