+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Early Pp module assembly intermediate of complex I | |||||||||
 マップデータ マップデータ | early Pp module assembly intermediate of complex I with assembly factors NDUFAF1 and CIA84 and cardiolipin remodeling enzyme tafazzin | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cardiolipin acyl-chain remodeling / 1-acylglycerophosphocholine O-acyltransferase activity / : / O-acyltransferase activity / inner mitochondrial membrane organization / phospholipid biosynthetic process / mitochondrial respirasome assembly / NADH:ubiquinone reductase (H+-translocating) / mitochondrial ATP synthesis coupled electron transport / : ...cardiolipin acyl-chain remodeling / 1-acylglycerophosphocholine O-acyltransferase activity / : / O-acyltransferase activity / inner mitochondrial membrane organization / phospholipid biosynthetic process / mitochondrial respirasome assembly / NADH:ubiquinone reductase (H+-translocating) / mitochondrial ATP synthesis coupled electron transport / : / mitochondrial membrane / mitochondrial inner membrane / oxidoreductase activity / mitochondrion 類似検索 - 分子機能 | |||||||||
| 生物種 |  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) | |||||||||
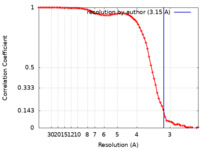
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||
 データ登録者 データ登録者 | Schiller J / Laube E / Vonck J / Zickermann V | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Insights into complex I assembly: Function of NDUFAF1 and a link with cardiolipin remodeling. 著者: Jonathan Schiller / Eike Laube / Ilka Wittig / Werner Kühlbrandt / Janet Vonck / Volker Zickermann /  要旨: Respiratory complex I is a ~1-MDa proton pump in mitochondria. Its structure has been revealed in great detail, but the structural basis of its assembly, in humans involving at least 15 assembly ...Respiratory complex I is a ~1-MDa proton pump in mitochondria. Its structure has been revealed in great detail, but the structural basis of its assembly, in humans involving at least 15 assembly factors, is essentially unknown. We determined cryo-electron microscopy structures of assembly intermediates associated with assembly factor NDUFAF1 in a yeast model system. Subunits ND2 and NDUFC2 together with assembly factors NDUFAF1 and CIA84 form the nucleation point of the NDUFAF1-dependent assembly pathway. Unexpectedly, the cardiolipin remodeling enzyme tafazzin is an integral component of this core complex. In a later intermediate, all 12 subunits of the proximal proton pump module have assembled. NDUFAF1 locks the central ND3 subunit in an assembly-competent conformation, and major rearrangements of central subunits are required for complex I maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14765.map.gz emd_14765.map.gz | 32.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14765-v30.xml emd-14765-v30.xml emd-14765.xml emd-14765.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14765_fsc.xml emd_14765_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14765.png emd_14765.png | 136.2 KB | ||
| その他 |  emd_14765_half_map_1.map.gz emd_14765_half_map_1.map.gz emd_14765_half_map_2.map.gz emd_14765_half_map_2.map.gz | 31.8 MB 31.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14765 http://ftp.pdbj.org/pub/emdb/structures/EMD-14765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14765 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14765_validation.pdf.gz emd_14765_validation.pdf.gz | 660.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14765_full_validation.pdf.gz emd_14765_full_validation.pdf.gz | 660 KB | 表示 | |
| XML形式データ |  emd_14765_validation.xml.gz emd_14765_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_14765_validation.cif.gz emd_14765_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14765 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14765 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14765.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14765.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | early Pp module assembly intermediate of complex I with assembly factors NDUFAF1 and CIA84 and cardiolipin remodeling enzyme tafazzin | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.20721 Å | ||||||||||||||||||||||||||||||||||||
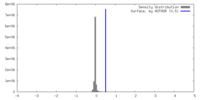
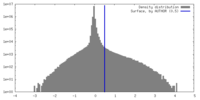
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_14765_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
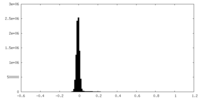
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14765_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
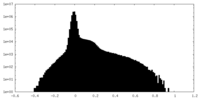
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Early Pp module assembly intermediate of complex I with assembly ...
| 全体 | 名称: Early Pp module assembly intermediate of complex I with assembly factors NDUFAF1 and CIA84 and cardiolipin remodeling enzyme tafazzin. |
|---|---|
| 要素 |
|
-超分子 #1: Early Pp module assembly intermediate of complex I with assembly ...
| 超分子 | 名称: Early Pp module assembly intermediate of complex I with assembly factors NDUFAF1 and CIA84 and cardiolipin remodeling enzyme tafazzin. タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
-分子 #1: NADH dehydrogenase subunit 2
| 分子 | 名称: NADH dehydrogenase subunit 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: NADH:ubiquinone reductase (H+-translocating) |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 53.381367 KDa |
| 配列 | 文字列: (FME)LILAIISLI TFVSMSKLSD NRAIIRLINI YLILVLVLDS FLYLLFLNNQ TYTVMGELLI FNSFTFYIDM LIYFIM IVI SSLYGYNLYN NNLYKTLFEP KKELIILFLI NILGALLIVH SNDFITLFVA IELQSYSIYL ITAIYNSSYK ASKASML YF ...文字列: (FME)LILAIISLI TFVSMSKLSD NRAIIRLINI YLILVLVLDS FLYLLFLNNQ TYTVMGELLI FNSFTFYIDM LIYFIM IVI SSLYGYNLYN NNLYKTLFEP KKELIILFLI NILGALLIVH SNDFITLFVA IELQSYSIYL ITAIYNSSYK ASKASML YF FMGGILSILI AYSINTYYSV LNSYTLHSLD SLIINTLDLN LILIALSLGL LFKIGIAPLH KWLISIYENT PILITIYI S LIPKISILSY LVLSNISINS LVISILAILT LLVGSVGGLL QIKIKRLLAF SGLTNAGYMM LLLLLNNNEF SYLYYITQY SISHLAIFMI IIFSIYYINY INNQYNPIIY VNQLKGLIHD NAYLVLSMAI VVFSFIGIPP LLGFFGKLNI LMSILNNGYY FISIVLIVA SLISALYYLY LLNVSIQDKN NILINSNETV SSVLSYILSS LIILITFGFI YNSLIIDIFN VYFN |
-分子 #2: Subunit NEBM of NADH:Ubiquinone Oxidoreductase (Complex I)
| 分子 | 名称: Subunit NEBM of NADH:Ubiquinone Oxidoreductase (Complex I) タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 8.059366 KDa |
| 配列 | 文字列: MALFTSLVGA SGLGFATKFL SNKIRLKPAG YYPLGYVFSG VAWAGLGLVL HNVHQHSLEV LEKKKTALSE QRTE |
-分子 #3: Complex I intermediate-associated protein 30-domain-containing protein
| 分子 | 名称: Complex I intermediate-associated protein 30-domain-containing protein タイプ: protein_or_peptide / ID: 3 / 詳細: C-terminal strep tag / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 31.894016 KDa |
| 組換発現 | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 配列 | 文字列: MSANPAIVRP TETTEQVLVN FTKPNSLETV LTKCDEELGG YSTVNLALER PTTGKPYGRF FGNLSLDLPK DNKMVTRSGF AMFRTLDQP SSMFKTNAWN WEQYRHLELR VRGDRRKYFV NVQSATPLAS DLYQHRLFIQ TPGEWETVVI PIDDFILTNK G VVQEQMAM ...文字列: MSANPAIVRP TETTEQVLVN FTKPNSLETV LTKCDEELGG YSTVNLALER PTTGKPYGRF FGNLSLDLPK DNKMVTRSGF AMFRTLDQP SSMFKTNAWN WEQYRHLELR VRGDRRKYFV NVQSATPLAS DLYQHRLFIQ TPGEWETVVI PIDDFILTNK G VVQEQMAM DTANVYTVGI GLIDRQYGPY NLDIEYIKAV AHPPLEFKPK KEYEVEKETI LLTPGQPMEL GKGKVKELEE NL YFQGAEA AAKEAAAKAW SHPQFEKGGG SGGGSGGSAW SHPQFEK |
-分子 #4: complex I assembly factor CIA84
| 分子 | 名称: complex I assembly factor CIA84 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 97.098469 KDa |
| 配列 | 文字列: MPKNALLRSA RQVAISRVFA TSRASHVVSH APILASVRPR SNPAPYRRNF SSSRALRNDY GLDTAERSLK ESLVPFNGAP VDRKVVRDQ LMELISVSPG QVFPISVIPV VKSAYYELFR ENERVLSAGD TKTLFGAVAG NNPEDVQDLP FVLAVYHQAE Q AAETNRDS ...文字列: MPKNALLRSA RQVAISRVFA TSRASHVVSH APILASVRPR SNPAPYRRNF SSSRALRNDY GLDTAERSLK ESLVPFNGAP VDRKVVRDQ LMELISVSPG QVFPISVIPV VKSAYYELFR ENERVLSAGD TKTLFGAVAG NNPEDVQDLP FVLAVYHQAE Q AAETNRDS RDNILLLGKY FLFQDRLDNF WKLLEAQIKT HDDVDAGFVK QLLELISVDP HLTLGNVARV LQLKTDNHVS SS DELRNAL SATLEQLYYK ENEGSEFFLS LVENHILDSK DFTPSDSVVA MILNTCVNEG REDLGQSVLR NVVSRVGNLS PGQ EDPQNC WGFWSSVAMD LHGSKTDVKA FISRLEALPH RTKATWDILI RYAVFKADLA GRNDLLQVRA LLAEMQKVGF EPDA ETYFD AYRSSKSIKP DVVHLFEAEL DIEKDTSIFA IEMDKALKNH DTLEALSIFY ESFEQGAQWE NKRLHMEAMT ELLIQ YAGL NDTSVADILQ LVQRIEPICA QGRIPYSAET AIAQNVLQRH SDTANFYTFM NRQYGNTADK VTKQDPQIRP HTYQVI HDY IYSCESERAD LAWEMYGLLH KFYVVPFADY YKAIKFFAQD VKRQDYALLT FQQIRKNHDL HGQPAATSEM VAFLFHE FA KTKYKRGIKR LHEVVALETS FDVNRDVLNE MMAAYVSVED LNRVQDCWAQ LQQLPPSIGA NNRSVDVLLS YFKDNIHY T ERTWQGIPEF GLLPTLENYE QYLINNCRTG NYRRALEITK NMEIDSGLKP TAKIIAAVYN YTFTEQRKLE VEQWAEKAH PEMWLELKEG DKLKSLCLPA NSDNDNVESL LKQASADMDE EMSGGIVKVE SV |
-分子 #5: Tafazzin family protein
| 分子 | 名称: Tafazzin family protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) / 株: CLIB 122 / E 150 Yarrowia lipolytica (酵母) / 株: CLIB 122 / E 150 |
| 分子量 | 理論値: 42.835367 KDa |
| 配列 | 文字列: MSFRNVSLRG SQLLGKLDSR GWGWYVAKKW NIGLVYTMCK VFLRCKKVDI KGLDNLLEAH RQARLEGRGL LTVMNHTSVL DDPVVWGML PNDNGWIPYL MRWATGAKDI CYKNKLYSLF FGAGQVLPIT RFGIGGPFQP GMDMCVRLLN PNNKIKYSAK Y TPYLVHTN ...文字列: MSFRNVSLRG SQLLGKLDSR GWGWYVAKKW NIGLVYTMCK VFLRCKKVDI KGLDNLLEAH RQARLEGRGL LTVMNHTSVL DDPVVWGML PNDNGWIPYL MRWATGAKDI CYKNKLYSLF FGAGQVLPIT RFGIGGPFQP GMDMCVRLLN PNNKIKYSAK Y TPYLVHTN ATSYPFWRES NWVHFFPEGY VHQALEPHEG TMRYFRWGTS RAVLEPVTPP IIVPMFSHGL QKVFQEIPKG YE MEGNNTN KDRTISIRIG EPISETTVAG FRNEWINLCH KENVGLNAET MPDVLKNGQE AKDLRSKVAA YLREEVEKLR LTV PNMNPE LPEFKEPEFW SDIDKVHKGV YNHRGKVRML RNPTKGLIEV VEANKD |
-分子 #6: CARDIOLIPIN
| 分子 | 名称: CARDIOLIPIN / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CDL |
|---|---|
| 分子量 | 理論値: 1.464043 KDa |
| Chemical component information |  ChemComp-CDL: |
-分子 #7: Lauryl Maltose Neopentyl Glycol
| 分子 | 名称: Lauryl Maltose Neopentyl Glycol / タイプ: ligand / ID: 7 / コピー数: 1 / 式: LMN |
|---|---|
| 分子量 | 理論値: 1.005188 KDa |
| Chemical component information |  ChemComp-AV0: |
-分子 #8: DIUNDECYL PHOSPHATIDYL CHOLINE
| 分子 | 名称: DIUNDECYL PHOSPHATIDYL CHOLINE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: PLC |
|---|---|
| 分子量 | 理論値: 622.834 Da |
| Chemical component information |  ChemComp-PLC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 276 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | The sample was a mixture of assembly intermediates associated with the assembly factor NDUFAF1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6229 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)