+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1442 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The role of the kinesin-13 neck in microtubule depolymerization. | |||||||||
 マップデータ マップデータ | A realspace map of kinesin-13-bound, 15 protofilament microtubules, calculated from averaged layer line data using Fourier-Bessel synthesis | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Moores CA / Hekmat-Nejad M / Sakowicz R / Milligan RA | |||||||||
 引用 引用 |  ジャーナル: Cell Cycle / 年: 2006 ジャーナル: Cell Cycle / 年: 2006タイトル: The role of the kinesin-13 neck in microtubule depolymerization. 著者: Carolyn A Moores / Jeremy Cooper / Mike Wagenbach / Yulia Ovechkina / Linda Wordeman / Ronald A Milligan /  要旨: To ensure genetic integrity, replicated chromosomes must be accurately distributed to daughter cells-a process that is accomplished on the microtubule spindle. Kinesin-13 motors play an essential ...To ensure genetic integrity, replicated chromosomes must be accurately distributed to daughter cells-a process that is accomplished on the microtubule spindle. Kinesin-13 motors play an essential role in this process by performing regulated microtubule depolymerization. We set out to dissect the depolymerization mechanism of these kinesins, and in particular, the role of their conserved neck sequence. We used a monomeric kinesin-13 MCAK, consisting of the neck and motor core, which has strong depolymerizing activity. In the presence of a non-hydrolysable ATP analogue, this construct induced formation of rings around microtubules. The rings are built from tubulin protofilaments that are bent by the kinesin-13 motor engaged at the ATP-binding step of its ATPase cycle. Our data suggest that the ring-microtubule interaction is mediated by the neck and support the idea of a role for the kinesin-13 neck in depolymerization efficiency, acting by optimizing release of tubulin from microtubule ends. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1442.map.gz emd_1442.map.gz | 476.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1442-v30.xml emd-1442-v30.xml emd-1442.xml emd-1442.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1442.gif 1442.gif | 109.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1442 http://ftp.pdbj.org/pub/emdb/structures/EMD-1442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1442 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1442_validation.pdf.gz emd_1442_validation.pdf.gz | 274.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1442_full_validation.pdf.gz emd_1442_full_validation.pdf.gz | 273.8 KB | 表示 | |
| XML形式データ |  emd_1442_validation.xml.gz emd_1442_validation.xml.gz | 3.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1442 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1442 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1442.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1442.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A realspace map of kinesin-13-bound, 15 protofilament microtubules, calculated from averaged layer line data using Fourier-Bessel synthesis | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.97 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
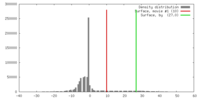
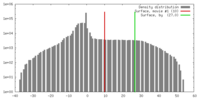
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Malarial kinesin-13 motor core ADPAlF
| 全体 | 名称: Malarial kinesin-13 motor core ADPAlF |
|---|---|
| 要素 |
|
-超分子 #1000: Malarial kinesin-13 motor core ADPAlF
| 超分子 | 名称: Malarial kinesin-13 motor core ADPAlF / タイプ: sample / ID: 1000 / 詳細: Molecular weight of kinesin motor core is 40kD / 集合状態: monomeric kinesin binds to each tubulin dimer / Number unique components: 2 |
|---|
-分子 #1: kinesin-13 motor core
| 分子 | 名称: kinesin-13 motor core / タイプ: protein_or_peptide / ID: 1 / Name.synonym: pKinI / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 40 KDa / 理論値: 40 KDa |
| 組換発現 | 生物種:  |
-分子 #2: tubulin
| 分子 | 名称: tubulin / タイプ: protein_or_peptide / ID: 2 / 集合状態: helical polymer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 100 KDa / 理論値: 100 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 詳細: 2mM ADP, 8mM NaF, 2mM AlCl3, 20mM Pipes, 2mM MgCl2, 1mM EGTA, 1mM DTT |
| グリッド | 詳細: 400 mesh quantifoil grid 2/2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: plunger. vitrification performed in humidified cold room 手法: blot for 3-4s before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 95 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at |
| 日付 | 2002年8月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: PERKIN ELMER / デジタル化 - サンプリング間隔: 20 µm / 実像数: 50 / 平均電子線量: 10 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.95 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダー: side entry, nitrogen cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Phoelix / 詳細: Final map calculated from 53 moire repeats |
|---|---|
| CTF補正 | 詳細: each microtubule |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)