+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bovine complex I in the presence of IM1761092, deactive class v (Consensus) | |||||||||
 マップデータ マップデータ | Consensus map globally sharpened | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.38 Å | |||||||||
 データ登録者 データ登録者 | Bridges HR / Blaza JN / Yin Z / Chung I / Hirst J | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structural basis of mammalian respiratory complex I inhibition by medicinal biguanides. 著者: Hannah R Bridges / James N Blaza / Zhan Yin / Injae Chung / Michael N Pollak / Judy Hirst /   要旨: The molecular mode of action of biguanides, including the drug metformin, which is widely used in the treatment of diabetes, is incompletely characterized. Here, we define the inhibitory drug-target ...The molecular mode of action of biguanides, including the drug metformin, which is widely used in the treatment of diabetes, is incompletely characterized. Here, we define the inhibitory drug-target interaction(s) of a model biguanide with mammalian respiratory complex I by combining cryo-electron microscopy and enzyme kinetics. We interpret these data to explain the selectivity of biguanide binding to different enzyme states. The primary inhibitory site is in an amphipathic region of the quinone-binding channel, and an additional binding site is in a pocket on the intermembrane-space side of the enzyme. An independent local chaotropic interaction, not previously described for any drug, displaces a portion of a key helix in the membrane domain. Our data provide a structural basis for biguanide action and enable the rational design of medicinal biguanides. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14293.map.gz emd_14293.map.gz | 1007.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14293-v30.xml emd-14293-v30.xml emd-14293.xml emd-14293.xml | 22.8 KB 22.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14293_fsc.xml emd_14293_fsc.xml | 23.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14293.png emd_14293.png | 101.1 KB | ||
| マスクデータ |  emd_14293_msk_1.map emd_14293_msk_1.map | 1.1 GB |  マスクマップ マスクマップ | |
| その他 |  emd_14293_additional_1.map.gz emd_14293_additional_1.map.gz emd_14293_half_map_1.map.gz emd_14293_half_map_1.map.gz emd_14293_half_map_2.map.gz emd_14293_half_map_2.map.gz | 1009.3 MB 892.3 MB 892.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14293 http://ftp.pdbj.org/pub/emdb/structures/EMD-14293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14293 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14293_validation.pdf.gz emd_14293_validation.pdf.gz | 917.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14293_full_validation.pdf.gz emd_14293_full_validation.pdf.gz | 917.3 KB | 表示 | |
| XML形式データ |  emd_14293_validation.xml.gz emd_14293_validation.xml.gz | 30.9 KB | 表示 | |
| CIF形式データ |  emd_14293_validation.cif.gz emd_14293_validation.cif.gz | 41.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14293 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14293 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14293 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14293 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14293.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14293.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus map globally sharpened | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.731 Å | ||||||||||||||||||||||||||||||||||||
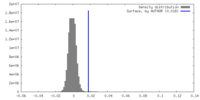
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14293_msk_1.map emd_14293_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
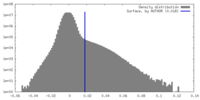
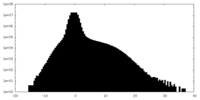
| 密度ヒストグラム |
-追加マップ: Consensus map locally sharpened
| ファイル | emd_14293_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus map locally sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
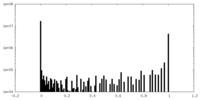
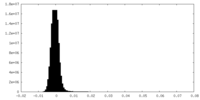
| 密度ヒストグラム |
-ハーフマップ: Consensus halfmap1
| ファイル | emd_14293_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus halfmap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
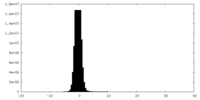
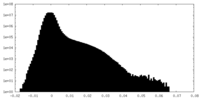
| 密度ヒストグラム |
-ハーフマップ: Consensus halfmap2
| ファイル | emd_14293_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus halfmap2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
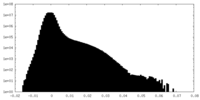
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NADH Ubiquinone oxidoreductase (Complex I)
| 全体 | 名称: NADH Ubiquinone oxidoreductase (Complex I) |
|---|---|
| 要素 |
|
-超分子 #1: NADH Ubiquinone oxidoreductase (Complex I)
| 超分子 | 名称: NADH Ubiquinone oxidoreductase (Complex I) / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#45 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.14 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R0.6/1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR 詳細: Grid also covalently modified by peg-thiol for 48 hours in a nitrogen atmosphere. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)