+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Abortive infection DNA polymerase AbiK from Lactococcus lactis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA polymerase / protein-primed DNA synthesis / template-independent DNA synthesis / abortive infection / ANTIVIRAL PROTEIN | |||||||||
| 機能・相同性 | Reverse transcriptase domain / Reverse transcriptase (RNA-dependent DNA polymerase) / Reverse transcriptase (RT) catalytic domain profile. / DNA/RNA polymerase superfamily / metal ion binding / AbiK 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Lactococcus lactis (乳酸菌) / Lactococcus lactis (乳酸菌) /  | |||||||||
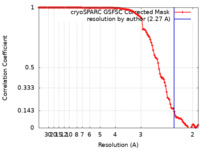
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.27 Å | |||||||||
 データ登録者 データ登録者 | Figiel M / Nowotny M / Gapinska M / Czarnocki-Cieciura M / Zajko W | |||||||||
| 資金援助 |  ポーランド, European Union, 2件 ポーランド, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Mechanism of protein-primed template-independent DNA synthesis by Abi polymerases. 著者: Małgorzata Figiel / Marta Gapińska / Mariusz Czarnocki-Cieciura / Weronika Zajko / Małgorzata Sroka / Krzysztof Skowronek / Marcin Nowotny 要旨: Abortive infection (Abi) is a bacterial antiphage defense strategy involving suicide of the infected cell. Some Abi pathways involve polymerases that are related to reverse transcriptases. They are ...Abortive infection (Abi) is a bacterial antiphage defense strategy involving suicide of the infected cell. Some Abi pathways involve polymerases that are related to reverse transcriptases. They are unique in the way they combine the ability to synthesize DNA in a template-independent manner with protein priming. Here, we report crystal and cryo-electron microscopy structures of two Abi polymerases: AbiK and Abi-P2. Both proteins adopt a bilobal structure with an RT-like domain that comprises palm and fingers subdomains and a unique helical domain. AbiK and Abi-P2 adopt a hexameric and trimeric configuration, respectively, which is unprecedented for reverse transcriptases. Biochemical experiments showed that the formation of these oligomers is required for the DNA polymerization activity. The structure of the AbiK-DNA covalent adduct visualized interactions between the 3' end of DNA and the active site and covalent attachment of the 5' end of DNA to a tyrosine residue used for protein priming. Our data reveal a structural basis of the mechanism of highly unusual template-independent protein-priming polymerases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14220.map.gz emd_14220.map.gz | 5.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14220-v30.xml emd-14220-v30.xml emd-14220.xml emd-14220.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14220_fsc.xml emd_14220_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14220.png emd_14220.png | 167.4 KB | ||
| マスクデータ |  emd_14220_msk_1.map emd_14220_msk_1.map | 149.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14220.cif.gz emd-14220.cif.gz | 6.5 KB | ||
| その他 |  emd_14220_half_map_1.map.gz emd_14220_half_map_1.map.gz emd_14220_half_map_2.map.gz emd_14220_half_map_2.map.gz | 139.1 MB 139.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14220 http://ftp.pdbj.org/pub/emdb/structures/EMD-14220 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14220 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14220 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14220_validation.pdf.gz emd_14220_validation.pdf.gz | 753 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14220_full_validation.pdf.gz emd_14220_full_validation.pdf.gz | 752.6 KB | 表示 | |
| XML形式データ |  emd_14220_validation.xml.gz emd_14220_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_14220_validation.cif.gz emd_14220_validation.cif.gz | 25.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14220 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14220 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14220 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14220 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14220.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14220.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14220_msk_1.map emd_14220_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14220_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14220_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homohexamer of AbiK with single-stranded DNA covalently attached ...
| 全体 | 名称: Homohexamer of AbiK with single-stranded DNA covalently attached to each of the subunits |
|---|---|
| 要素 |
|
-超分子 #1: Homohexamer of AbiK with single-stranded DNA covalently attached ...
| 超分子 | 名称: Homohexamer of AbiK with single-stranded DNA covalently attached to each of the subunits タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 420 KDa |
-超分子 #2: Abortive infection protein AbiK
| 超分子 | 名称: Abortive infection protein AbiK / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
-超分子 #3: DNA (5'-D(*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*C)-3')
| 超分子 | 名称: DNA (5'-D(*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*C)-3') / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: AbiK
| 分子 | 名称: AbiK / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
| 分子量 | 理論値: 71.713086 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMKKEFTEL YDFIFDPIFL VRYGYYDRSI KNKKMNTAKV ELDNE(PTR)GKSD SFYFKVFNME SFADYLRSHD LKTHFN GKK PLSTDPVYFN IPKNIEARRQ YKMPNLYSYM ALNYYICDNK KEFIEVFIDN KFSTSKFFNQ LNFDYPKTQE ITQTLLY GG ...文字列: GSMKKEFTEL YDFIFDPIFL VRYGYYDRSI KNKKMNTAKV ELDNE(PTR)GKSD SFYFKVFNME SFADYLRSHD LKTHFN GKK PLSTDPVYFN IPKNIEARRQ YKMPNLYSYM ALNYYICDNK KEFIEVFIDN KFSTSKFFNQ LNFDYPKTQE ITQTLLY GG IKKLHLDLSN FYHTLYTHSI PWMIDGKSAS KQNRKKGFSN TLDTLITACQ YDETHGIPTG NLLSRIITEL YMCHFDKQ M EYKKFVYSRY VDDFIFPFTF ENEKQEFLNE FNLICRENNL IINDNKTKVD NFPFVDKSSK SDIFSFFENI TSTNSNDKW IKEISNFIDY CVNEEHLGNK GAIKCIFPVI TNTLKQKKVD TKNIDNIFSK RNMVTNFNVF EKILDLSLKD SRLTNKFLTF FENINEFGF SSLSASNIVK KYFSNNSKGL KEKIDHYRKN NFNQELYQIL LYMVVFEIDD LLNQEELLNL IDLNIDDYSL I LGTILYLK NSSYKLEKLL KKIDQLFINT HANYDVKTSR MAEKLWLFRY FFYFLNCKNI FSQKEINSYC QSQNYNSGQN GY QTELNWN YIKGQGKDLR ANNFFNELIV KEVWLISCGE NEDFKYLN UniProtKB: AbiK |
-分子 #2: DNA (5'-D(*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*C)-3')
| 分子 | 名称: DNA (5'-D(*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*CP*C)-3') / タイプ: dna / ID: 2 / コピー数: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 3.425224 KDa |
| 配列 | 文字列: (DC)(DC)(DC)(DC)(DC)(DC)(DC)(DC)(DC)(DC) (DC)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)