+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13046 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Brr2 in complex with Jab1/MPN and C9ORF78 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mRNA Splicing / Spliceosomal Assembly / Splicing Regulation / Brr2 Helicase / SPLICING | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of homologous chromosome segregation / cis assembly of pre-catalytic spliceosome / spliceosome conformational change to release U4 (or U4atac) and U1 (or U11) / U2-type catalytic step 1 spliceosome / RNA splicing, via transesterification reactions / mRNA cis splicing, via spliceosome / U2-type precatalytic spliceosome / U2-type catalytic step 2 spliceosome / K63-linked polyubiquitin modification-dependent protein binding / mRNA Splicing - Minor Pathway ...regulation of homologous chromosome segregation / cis assembly of pre-catalytic spliceosome / spliceosome conformational change to release U4 (or U4atac) and U1 (or U11) / U2-type catalytic step 1 spliceosome / RNA splicing, via transesterification reactions / mRNA cis splicing, via spliceosome / U2-type precatalytic spliceosome / U2-type catalytic step 2 spliceosome / K63-linked polyubiquitin modification-dependent protein binding / mRNA Splicing - Minor Pathway / chromosome, centromeric region / spliceosomal tri-snRNP complex assembly / U5 snRNA binding / U5 snRNP / U2 snRNA binding / U6 snRNA binding / pre-mRNA intronic binding / U1 snRNA binding / U4/U6 x U5 tri-snRNP complex / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / RNA splicing / helicase activity / chromosome segregation / spliceosomal complex / mRNA processing / mRNA splicing, via spliceosome / osteoblast differentiation / cellular response to tumor necrosis factor / cellular response to lipopolysaccharide / RNA helicase activity / RNA helicase / nuclear speck / ATP hydrolysis activity / RNA binding / nucleoplasm / ATP binding / identical protein binding / membrane / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
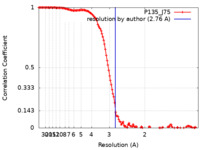
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.76 Å | |||||||||
 データ登録者 データ登録者 | Bergfort A / Hilal T | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: The intrinsically disordered TSSC4 protein acts as a helicase inhibitor, placeholder and multi-interaction coordinator during snRNP assembly and recycling. 著者: Alexandra Bergfort / Tarek Hilal / Benno Kuropka / İbrahim Avşar Ilik / Gert Weber / Tuğçe Aktaş / Christian Freund / Markus C Wahl /  要旨: Biogenesis of spliceosomal small nuclear ribonucleoproteins (snRNPs) and their recycling after splicing require numerous assembly/recycling factors whose modes of action are often poorly understood. ...Biogenesis of spliceosomal small nuclear ribonucleoproteins (snRNPs) and their recycling after splicing require numerous assembly/recycling factors whose modes of action are often poorly understood. The intrinsically disordered TSSC4 protein has been identified as a nuclear-localized U5 snRNP and U4/U6-U5 tri-snRNP assembly/recycling factor, but how TSSC4's intrinsic disorder supports TSSC4 functions remains unknown. Using diverse interaction assays and cryogenic electron microscopy-based structural analysis, we show that TSSC4 employs four conserved, non-contiguous regions to bind the PRPF8 Jab1/MPN domain and the SNRNP200 helicase at functionally important sites. It thereby inhibits SNRNP200 helicase activity, spatially aligns the proteins, coordinates formation of a U5 sub-module and transiently blocks premature interaction of SNRNP200 with at least three other spliceosomal factors. Guided by the structure, we designed a TSSC4 variant that lacks stable binding to the PRPF8 Jab1/MPN domain or SNRNP200 in vitro. Comparative immunoprecipitation/mass spectrometry from HEK293 nuclear extract revealed distinct interaction profiles of wild type TSSC4 and the variant deficient in PRPF8/SNRNP200 binding with snRNP proteins, other spliceosomal proteins as well as snRNP assembly/recycling factors and chaperones. Our findings elucidate molecular strategies employed by an intrinsically disordered protein to promote snRNP assembly, and suggest multiple TSSC4-dependent stages during snRNP assembly/recycling. #1:  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019タイトル: Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix. 著者: Liebschner D / Afonine PV / Baker ML / Bunkoczi G / Chen VB / Croll TI / Hintze B / Hung LW / Jain S / McCoy AJ / Moriarty NW / Oeffner RD / Poon BK / Prisant MG / Read RJ / Richardson JS / ...著者: Liebschner D / Afonine PV / Baker ML / Bunkoczi G / Chen VB / Croll TI / Hintze B / Hung LW / Jain S / McCoy AJ / Moriarty NW / Oeffner RD / Poon BK / Prisant MG / Read RJ / Richardson JS / Richardson DC / Sammito MD / Sobolev OV / Stockwell DH / Terwilliger TC / Urzhumtsev AG / Videau LL / Williams CJ / Adams PD | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13046.map.gz emd_13046.map.gz | 41.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13046-v30.xml emd-13046-v30.xml emd-13046.xml emd-13046.xml | 21.5 KB 21.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13046_fsc.xml emd_13046_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13046.png emd_13046.png | 101.7 KB | ||
| Filedesc metadata |  emd-13046.cif.gz emd-13046.cif.gz | 8.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13046 http://ftp.pdbj.org/pub/emdb/structures/EMD-13046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13046 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13046_validation.pdf.gz emd_13046_validation.pdf.gz | 467.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13046_full_validation.pdf.gz emd_13046_full_validation.pdf.gz | 467.3 KB | 表示 | |
| XML形式データ |  emd_13046_validation.xml.gz emd_13046_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_13046_validation.cif.gz emd_13046_validation.cif.gz | 18.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13046 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13046 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13046.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13046.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.657 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Trimeric complex of Brr2, the Jab1/MPN domain of Prp8 and a N-ter...
| 全体 | 名称: Trimeric complex of Brr2, the Jab1/MPN domain of Prp8 and a N-terminal fragment of C9ORF78 |
|---|---|
| 要素 |
|
-超分子 #1: Trimeric complex of Brr2, the Jab1/MPN domain of Prp8 and a N-ter...
| 超分子 | 名称: Trimeric complex of Brr2, the Jab1/MPN domain of Prp8 and a N-terminal fragment of C9ORF78 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: U5 small nuclear ribonucleoprotein 200 kDa helicase (Brr2)
| 超分子 | 名称: U5 small nuclear ribonucleoprotein 200 kDa helicase (Brr2) タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Telomere length and silencing protein 1 homolog (C9ORF78)
| 超分子 | 名称: Telomere length and silencing protein 1 homolog (C9ORF78) タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: Pre-mRNA-processing-splicing factor 8 (Prp8) Jab1/MPN Domain
| 超分子 | 名称: Pre-mRNA-processing-splicing factor 8 (Prp8) Jab1/MPN Domain タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: U5 small nuclear ribonucleoprotein 200 kDa helicase
| 分子 | 名称: U5 small nuclear ribonucleoprotein 200 kDa helicase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 198.785797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAEFDLDQGG EALAPRQVLD LEDLVFTQGS HFMANKRCQL PDGSFRRQRK GYEEVHVPAL KPKPFGSEEQ LLPVEKLPKY AQAGFEGFK TLNRIQSKLY RAALETDENL LLCAPTGAGK TNVALMCMLR EIGKHINMDG TINVDDFKII YIAPMRSLVQ E MVGSFGKR ...文字列: GAEFDLDQGG EALAPRQVLD LEDLVFTQGS HFMANKRCQL PDGSFRRQRK GYEEVHVPAL KPKPFGSEEQ LLPVEKLPKY AQAGFEGFK TLNRIQSKLY RAALETDENL LLCAPTGAGK TNVALMCMLR EIGKHINMDG TINVDDFKII YIAPMRSLVQ E MVGSFGKR LATYGITVAE LTGDHQLCKE EISATQIIVC TPEKWDIITR KGGERTYTQL VRLIILDEIH LLHDDRGPVL EA LVARAIR NIEMTQEDVR LIGLSATLPN YEDVATFLRV DPAKGLFYFD NSFRPVPLEQ TYVGITEKKA IKRFQIMNEI VYE KIMEHA GKNQVLVFVH SRKETGKTAR AIRDMCLEKD TLGLFLREGS ASTEVLRTEA EQCKNLELKD LLPYGFAIHH AGMT RVDRT LVEDLFADKH IQVLVSTATL AWGVNLPAHT VIIKGTQVYS PEKGRWTELG ALDILQMLGR AGRPQYDTKG EGILI TSHG ELQYYLSLLN QQLPIESQMV SKLPDMLNAE IVLGNVQNAK DAVNWLGYAY LYIRMLRSPT LYGISHDDLK GDPLLD QRR LDLVHTAALM LDKNNLVKYD KKTGNFQVTE LGRIASHYYI TNDTVQTYNQ LLKPTLSEIE LFRVFSLSSE FKNITVR EE EKLELQKLLE RVPIPVKESI EEPSAKINVL LQAFISQLKL EGFALMADMV YVTQSAGRLM RAIFEIVLNR GWAQLTDK T LNLCKMIDKR MWQSMCPLRQ FRKLPEEVVK KIEKKNFPFE RLYDLNHNEI GELIRMPKMG KTIHKYVHLF PKLELSVHL QPITRSTLKV ELTITPDFQW DEKVHGSSEA FWILVEDVDS EVILHHEYFL LKAKYAQDEH LITFFVPVFE PLPPQYFIRV VSDRWLSCE TQLPVSFRHL ILPEKYPPPT ELLDLQPLPV SALRNSAFES LYQDKFPFFN PIQTQVFNTV YNSDDNVFVG A PTGSGKTI CAEFAILRML LQSSEGRCVY ITPMEALAEQ VYMDWYEKFQ DRLNKKVVLL TGETSTDLKL LGKGNIIIST PE KWDILSR RWKQRKNVQN INLFVVDEVH LIGGENGPVL EVICSRMRYI SSQIERPIRI VALSSSLSNA KDVAHWLGCS ATS TFNFHP NVRPVPLELH IQGFNISHTQ TRLLSMAKPV YHAITKHSPK KPVIVFVPSR KQTRLTAIDI LTTCAADIQR QRFL HCTEK DLIPYLEKLS DSTLKETLLN GVGYLHEGLS PMERRLVEQL FSSGAIQVVV ASRSLCWGMN VAAHLVIIMD TQYYN GKIH AYVDYPIYDV LQMVGHANRP LQDDEGRCVI MCQGSKKDFF KKFLYEPLPV ESHLDHCMHD HFNAEIVTKT IENKQD AVD YLTWTFLYRR MTQNPNYYNL QGISHRHLSD HLSELVEQTL SDLEQSKCIS IEDEMDVAPL NLGMIAAYYY INYTTIE LF SMSLNAKTKV RGLIEIISNA AEYENIPIRH HEDNLLRQLA QKVPHKLNNP KFNDPHVKTN LLLQAHLSRM QLSAELQS D TEEILSKAIR LIQACVDVLS SNGWLSPALA AMELAQMVTQ AMWSKDSYLK QLPHFTSEHI KRCTDKGVES VFDIMEMED EERNALLQLT DSQIADVARF CNRYPNIELS YEVVDKDSIR SGGPVVVLVQ LEREEEVTGP VIAPLFPQKR EEGWWVVIGD AKSNSLISI KRLTLQQKAK VKLDFVAPAT GAHNYTLYFM SDAYMGCDQE YKFSVDVKEA UniProtKB: U5 small nuclear ribonucleoprotein 200 kDa helicase |
-分子 #2: Telomere length and silencing protein 1 homolog
| 分子 | 名称: Telomere length and silencing protein 1 homolog / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34.081516 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAMAMPVVRK IFRRRRGDSE SEEDEQDSEE VRLKLEETRE VQNLRKRPNG VSAVALLVGE KVQEETTLVD DPFQMKTGGM VDMKKLKER GKDKISEEED LHLGTSFSAE TNRRDEDADM MKYIETELKK RKGIVEHEEQ KVKPKNAEDC LYELPENIRV S SAKKTEEM ...文字列: GAMAMPVVRK IFRRRRGDSE SEEDEQDSEE VRLKLEETRE VQNLRKRPNG VSAVALLVGE KVQEETTLVD DPFQMKTGGM VDMKKLKER GKDKISEEED LHLGTSFSAE TNRRDEDADM MKYIETELKK RKGIVEHEEQ KVKPKNAEDC LYELPENIRV S SAKKTEEM LSNQMLSGIP EVDLGIDAKI KNIISTEDAK ARLLAEQQNK KKDSETSFVP TNMAVNYVQH NRFYHEELNA PI RRNKEEP KARPLRVGDT EKPEPERSPP NRKRPANEKA TDDYHYEKFK KMNRRY UniProtKB: Splicing factor C9orf78 |
-分子 #3: Pre-mRNA-processing-splicing factor 8
| 分子 | 名称: Pre-mRNA-processing-splicing factor 8 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.860984 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGSMTQTF SSKTEWRVRA ISAANLHLRT NHIYVSSDDI KETGYTYILP KNVLKKFICI SDLRAQIAGY LYGVSPPDNP QVKEIRCIV MVPQWGTHQT VHLPGQLPQH EYLKEMEPLG WIHTQPNESP QLSPQDVTTH AKIMADNPSW DGEKTIIITC S FTPGSCTL ...文字列: GPLGSMTQTF SSKTEWRVRA ISAANLHLRT NHIYVSSDDI KETGYTYILP KNVLKKFICI SDLRAQIAGY LYGVSPPDNP QVKEIRCIV MVPQWGTHQT VHLPGQLPQH EYLKEMEPLG WIHTQPNESP QLSPQDVTTH AKIMADNPSW DGEKTIIITC S FTPGSCTL TAYKLTPSGY EWGRQNTDKG NNPKGYLPSH YERVQMLLSD RFLGFFMVPA QSSWNYNFMG VRHDPNMKYE LQ LANPKEF YHEVHRPSHF LNFALLQEGE VYSADREDLY A UniProtKB: Pre-mRNA-processing-splicing factor 8 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 5160 / 平均露光時間: 40.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 102 / 当てはまり具合の基準: CC, R.m.s.d. Bonds / Angles | ||||||
| 得られたモデル |  PDB-7os2: |
 ムービー
ムービー コントローラー
コントローラー