[日本語] English
 万見
万見- EMDB-12263: structure of a POTRA-locked BAM complex in a lateral-open conformation -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12263 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | structure of a POTRA-locked BAM complex in a lateral-open conformation | ||||||||||||||||||||||||
 マップデータ マップデータ | Postprocessed masked map of a POTRA-locked BAM complex | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane / cell outer membrane / protein-macromolecule adaptor activity / cell adhesion / response to antibiotic / cell surface / identical protein binding / membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   | ||||||||||||||||||||||||
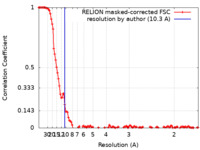
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.3 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Machin JM / Haysom SF | ||||||||||||||||||||||||
| 資金援助 |  英国, 英国,  ベルギー, 7件 ベルギー, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: The role of membrane destabilisation and protein dynamics in BAM catalysed OMP folding. 著者: Paul White / Samuel F Haysom / Matthew G Iadanza / Anna J Higgins / Jonathan M Machin / James M Whitehouse / Jim E Horne / Bob Schiffrin / Charlotte Carpenter-Platt / Antonio N Calabrese / ...著者: Paul White / Samuel F Haysom / Matthew G Iadanza / Anna J Higgins / Jonathan M Machin / James M Whitehouse / Jim E Horne / Bob Schiffrin / Charlotte Carpenter-Platt / Antonio N Calabrese / Kelly M Storek / Steven T Rutherford / David J Brockwell / Neil A Ranson / Sheena E Radford /   要旨: The folding of β-barrel outer membrane proteins (OMPs) in Gram-negative bacteria is catalysed by the β-barrel assembly machinery (BAM). How lateral opening in the β-barrel of the major subunit ...The folding of β-barrel outer membrane proteins (OMPs) in Gram-negative bacteria is catalysed by the β-barrel assembly machinery (BAM). How lateral opening in the β-barrel of the major subunit BamA assists in OMP folding, and the contribution of membrane disruption to BAM catalysis remain unresolved. Here, we use an anti-BamA monoclonal antibody fragment (Fab1) and two disulphide-crosslinked BAM variants (lid-locked (LL), and POTRA-5-locked (P5L)) to dissect these roles. Despite being lethal in vivo, we show that all complexes catalyse folding in vitro, albeit less efficiently than wild-type BAM. CryoEM reveals that while Fab1 and BAM-P5L trap an open-barrel state, BAM-LL contains a mixture of closed and contorted, partially-open structures. Finally, all three complexes globally destabilise the lipid bilayer, while BamA does not, revealing that the BAM lipoproteins are required for this function. Together the results provide insights into the role of BAM structure and lipid dynamics in OMP folding. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12263.map.gz emd_12263.map.gz | 154.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12263-v30.xml emd-12263-v30.xml emd-12263.xml emd-12263.xml | 25.8 KB 25.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12263_fsc.xml emd_12263_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12263.png emd_12263.png | 116.6 KB | ||
| マスクデータ |  emd_12263_msk_1.map emd_12263_msk_1.map | 166.4 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12263_half_map_1.map.gz emd_12263_half_map_1.map.gz emd_12263_half_map_2.map.gz emd_12263_half_map_2.map.gz | 131.5 MB 131.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12263 http://ftp.pdbj.org/pub/emdb/structures/EMD-12263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12263 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12263_validation.pdf.gz emd_12263_validation.pdf.gz | 469.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12263_full_validation.pdf.gz emd_12263_full_validation.pdf.gz | 468.6 KB | 表示 | |
| XML形式データ |  emd_12263_validation.xml.gz emd_12263_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_12263_validation.cif.gz emd_12263_validation.cif.gz | 25 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12263 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12263 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12263 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12263 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12263.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12263.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed masked map of a POTRA-locked BAM complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12263_msk_1.map emd_12263_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 1 from Refine3D (RELION 3.1)
| ファイル | emd_12263_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 1 from Refine3D (RELION 3.1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 2 from Refine3D (RELION 3.1)
| ファイル | emd_12263_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 2 from Refine3D (RELION 3.1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : POTRA-locked beta-barrel assembly machinery (BAM) complex
| 全体 | 名称: POTRA-locked beta-barrel assembly machinery (BAM) complex |
|---|---|
| 要素 |
|
-超分子 #1: POTRA-locked beta-barrel assembly machinery (BAM) complex
| 超分子 | 名称: POTRA-locked beta-barrel assembly machinery (BAM) complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: beta-barrel assembly machinery (BAM) complex (BamABCDE) with POTRA-lock in BamA (G393C G584C) and natural cysteines removed (C690S C700S) |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 203 KDa |
-分子 #1: Outer membrane protein assembly factor BamA
| 分子 | 名称: Outer membrane protein assembly factor BamA / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAMKKLLIAS LLFSSATVYG AEGFVVKDIH FEGLQRVAVG AALLSMPVRT GDTVNDEDIS NTIRALFATG NFEDVRVLRD GDTLLVQVKE RPTIASITFS GNKSVKDDML KQNLEASGVR VGESLDRTTI ADIEKGLEDF YYSVGKYSAS VKAVVTPLPR NRVDLKLVFQ ...文字列: MAMKKLLIAS LLFSSATVYG AEGFVVKDIH FEGLQRVAVG AALLSMPVRT GDTVNDEDIS NTIRALFATG NFEDVRVLRD GDTLLVQVKE RPTIASITFS GNKSVKDDML KQNLEASGVR VGESLDRTTI ADIEKGLEDF YYSVGKYSAS VKAVVTPLPR NRVDLKLVFQ EGVSAEIQQI NIVGNHAFTT DELISHFQLR DEVPWWNVVG DRKYQKQKLA GDLETLRSYY LDRGYARFNI DSTQVSLTPD KKGIYVTVNI TEGDQYKLSG VEVSGNLAGH SAEIEQLTKI EPGELYNGTK VTKMEDDIKK LLGRYGYAYP RVQSMPEIND ADKTVKLRVN VDAGNRFYVR KIRFEGNDTS KDAVLRREMR QMEGAWLGSD LVDQGKERLN RLGFFETVDT DTQRVPGSPD QVDVVYKVKE RNTGSFNFGI GYGTESGVSF QAGVQQDNWL GTGYAVGING TKNDYQTYAE LSVTNPYFTV DGVSLGGRLF YNDFQADDAD LSDYTNKSYG TDVTLGFPIN EYNSLRAGLG YVHNSLSNMQ PQVAMWRYLY SMGEHPSTSD QDNSFKTDDF TFNYGWTYNK LDRGYFPTDG SRVNLTGKVT IPGSDNEYYK VTLDTATYVP IDDDHKWVVL GRTRWGYGDG LGGKEMPFYE NFYAGGSSTV RGFQSNTIGP KAVYFPHQAS NYDPDYDYEC ATQDGAKDLC KSDDAVGGNA MAVASLEFIT PTPFISDKYA NSVRTSFFWD MGTVWDTNWD SSQYSGYPDY SDPSNIRMSA GIALQWMSPL GPLVFSYAQP FKKYDGDKAE QFQFNIGKTW |
-分子 #2: Outer membrane protein assembly factor BamB
| 分子 | 名称: Outer membrane protein assembly factor BamB / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALADN VVYAADRAGL VKALNADDGK EIWSVSLAEK DGWFSKEPAL LSGGVTVSGG HVYIGSEKAQ VYALNTSDGT VAWQTKVAGE ALSRPVVSDG LVLIHTSNGQ ...文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALADN VVYAADRAGL VKALNADDGK EIWSVSLAEK DGWFSKEPAL LSGGVTVSGG HVYIGSEKAQ VYALNTSDGT VAWQTKVAGE ALSRPVVSDG LVLIHTSNGQ LQALNEADGA VKWTVNLDMP SLSLRGESAP TTAFGAAVVG GDNGRVSAVL MEQGQMIWQQ RISQATGSTE IDRLSDVDTT PVVVNGVVFA LAYNGNLTAL DLRSGQIMWK RELGSVNDFI VDGNRIYLVD QNDRVMALTI DGGVTLWTQS DLLHRLLTSP VLYNGNLVVG DSEGYLHWIN VEDGRFVAQQ KVDSSGFQTE PVAADGKLLI QAKDGTVYSI TR |
-分子 #3: Outer membrane protein assembly factor BamC
| 分子 | 名称: Outer membrane protein assembly factor BamC / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPVT NGSGAVGKAL DIRPPAQPLA LVSGARTQFT GDTASLLVEN GRGNTLWPQV VSVLQAKNYT ITQRDDAGQT LTTDWVQWNR LDEDEQYRGR YQISVKPQGY ...文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPVT NGSGAVGKAL DIRPPAQPLA LVSGARTQFT GDTASLLVEN GRGNTLWPQV VSVLQAKNYT ITQRDDAGQT LTTDWVQWNR LDEDEQYRGR YQISVKPQGY QQAVTVKLLN LEQAGKPVAD AASMQRYSTE MMNVISAGLD KSATDAANAA QNRASTTMDV QSAADDTGLP MLVVRGPFNV VWQRLPAALE KVGMKVTDST RSQGNMAVTY KPLSDSDWQE LGASDPGLAS GDYKLQVGDL DNRSSLQFID PKGHTLTQSQ NDALVAVFQA AFSK |
-分子 #4: Outer membrane protein assembly factor BamD
| 分子 | 名称: Outer membrane protein assembly factor BamD / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQQ VQLDLIYAYY KNADLPLAQA AIDRFIRLNP THPNIDYVMY MRGLTNMALD DSALQGFFGV DRSDRDPQHA RAAFSDFSKL VRGYPNSQYT TDATKRLVFL ...文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQQ VQLDLIYAYY KNADLPLAQA AIDRFIRLNP THPNIDYVMY MRGLTNMALD DSALQGFFGV DRSDRDPQHA RAAFSDFSKL VRGYPNSQYT TDATKRLVFL KDRLAKYEYS VAEYYTERGA WVAVVNRVEG MLRDYPDTQA TRDALPLMEN AYRQMQMNAQ AEKVAKIIAA NSSNT |
-分子 #5: Outer membrane protein assembly factor BamE
| 分子 | 名称: Outer membrane protein assembly factor BamE / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MRCKTLTAAA AVLLMLTAGC STLERVVYRP DINQGNYLTA NDVSKIRVGM TQQQVAYALG TPLMSDPFGT NTWFYVFRQQ PGHEGVTQQT LTLTFNSSGV LTNIDNKPAL SGN |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: Glow discharge using GloqQube Plus, 30s, 60 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X