+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12099 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication | ||||||||||||||||||||||||
 マップデータ マップデータ | Helical fibril of foot and mouth disease virus RNA dependent RNA polymerase (3Dpol). Conformation N6 | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host chromatin organization / ribonucleoside triphosphate phosphatase activity / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / regulation of translation / channel activity / monoatomic ion transmembrane transport / clathrin-dependent endocytosis of virus by host cell / RNA helicase activity / viral protein processing ...symbiont-mediated perturbation of host chromatin organization / ribonucleoside triphosphate phosphatase activity / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / regulation of translation / channel activity / monoatomic ion transmembrane transport / clathrin-dependent endocytosis of virus by host cell / RNA helicase activity / viral protein processing / host cell endoplasmic reticulum membrane / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / proteolysis / RNA binding / ATP binding / metal ion binding / membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Foot-and-mouth disease virus (口蹄疫ウイルス) Foot-and-mouth disease virus (口蹄疫ウイルス) | ||||||||||||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 7.6 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Loundras EA / Streetley J / Herod MJ / Thompson R / Harris M / Bhella D / Stonehouse NJ | ||||||||||||||||||||||||
| 資金援助 |  英国, 7件 英国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for genome replication. 著者: Eleni-Anna Loundras / James Streetley / Morgan R Herod / Rebecca Thompson / Mark Harris / David Bhella / Nicola J Stonehouse /  要旨: Replication of many positive-sense RNA viruses occurs within intracellular membrane-associated compartments. These are thought to provide a favourable environment for replication to occur, ...Replication of many positive-sense RNA viruses occurs within intracellular membrane-associated compartments. These are thought to provide a favourable environment for replication to occur, concentrating essential viral structural and nonstructural components, as well as protecting these components from host-cell pathogen recognition and innate immune responses. However, the details of the molecular interactions and dynamics within these structures is very limited. One of the key components of the replication machinery is the RNA-dependent RNA polymerase, RdRp. This enzyme has been shown to form higher-order fibrils in vitro. Here, using the RdRp from foot-and-mouth disease virus (termed 3D), we report fibril structures, solved at ~7-9 Å resolution by cryo-EM, revealing multiple conformations of a flexible assembly. Fitting high-resolution coordinates led to the definition of potential intermolecular interactions. We employed mutagenesis using a sub-genomic replicon system to probe the importance of these interactions for replication. We use these data to propose models for the role of higher-order 3D complexes as a dynamic scaffold within which RNA replication can occur. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12099.map.gz emd_12099.map.gz | 5.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12099-v30.xml emd-12099-v30.xml emd-12099.xml emd-12099.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12099_fsc.xml emd_12099_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12099.png emd_12099.png | 188.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12099 http://ftp.pdbj.org/pub/emdb/structures/EMD-12099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12099 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12099_validation.pdf.gz emd_12099_validation.pdf.gz | 369.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12099_full_validation.pdf.gz emd_12099_full_validation.pdf.gz | 368.9 KB | 表示 | |
| XML形式データ |  emd_12099_validation.xml.gz emd_12099_validation.xml.gz | 9.8 KB | 表示 | |
| CIF形式データ |  emd_12099_validation.cif.gz emd_12099_validation.cif.gz | 12.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12099 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12099 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12099 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12099 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12099.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12099.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical fibril of foot and mouth disease virus RNA dependent RNA polymerase (3Dpol). Conformation N6 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
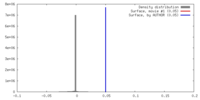
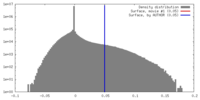
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Helical fibril of foot and mouth disease virus RNA dependent RNA ...
| 全体 | 名称: Helical fibril of foot and mouth disease virus RNA dependent RNA polymerase (3Dpol) |
|---|---|
| 要素 |
|
-超分子 #1: Helical fibril of foot and mouth disease virus RNA dependent RNA ...
| 超分子 | 名称: Helical fibril of foot and mouth disease virus RNA dependent RNA polymerase (3Dpol) タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus (口蹄疫ウイルス) Foot-and-mouth disease virus (口蹄疫ウイルス) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Specimen was loaded onto C-flat (1.2/1.3), 400 mesh holey carbon support films bearing a continuous carbon layer. Allowed to adsorb for one minute before blotting for 6-seconds and plunge freezing.. |
| 詳細 | Fibrils were formed by mixing FMDV 3DPol with a 13mer of poly-adenosine RNA, oligo-d(T) primer and UTP in the presence of 5mM glutaraldehyde. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 2 / 実像数: 5417 / 平均露光時間: 2.0 sec. / 平均電子線量: 108.0 e/Å2 詳細: The dose rate is 60 e/pix/s, giving a dose of 54 e/a2/second. We are taking a 2 second exposure, with 79 frames in total, giving 1.4 electrons/frame |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)