+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11707 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of B. subtilis ClpC (DWB mutant) hexamer bound to a substrate polypeptide | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報establishment of competence for transformation / cellular response to heat / ATP hydrolysis activity / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
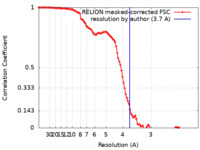
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Morreale FE / Meinhart A / Haselbach D / Clausen T | |||||||||
| 資金援助 |  オーストリア, 1件 オーストリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: BacPROTACs mediate targeted protein degradation in bacteria. 著者: Francesca E Morreale / Stefan Kleine / Julia Leodolter / Sabryna Junker / David M Hoi / Stepan Ovchinnikov / Anastasia Okun / Juliane Kley / Robert Kurzbauer / Lukas Junk / Somraj Guha / ...著者: Francesca E Morreale / Stefan Kleine / Julia Leodolter / Sabryna Junker / David M Hoi / Stepan Ovchinnikov / Anastasia Okun / Juliane Kley / Robert Kurzbauer / Lukas Junk / Somraj Guha / David Podlesainski / Uli Kazmaier / Guido Boehmelt / Harald Weinstabl / Klaus Rumpel / Volker M Schmiedel / Markus Hartl / David Haselbach / Anton Meinhart / Markus Kaiser / Tim Clausen /   要旨: Hijacking the cellular protein degradation system offers unique opportunities for drug discovery, as exemplified by proteolysis-targeting chimeras. Despite their great promise for medical chemistry, ...Hijacking the cellular protein degradation system offers unique opportunities for drug discovery, as exemplified by proteolysis-targeting chimeras. Despite their great promise for medical chemistry, so far, it has not been possible to reprogram the bacterial degradation machinery to interfere with microbial infections. Here, we develop small-molecule degraders, so-called BacPROTACs, that bind to the substrate receptor of the ClpC:ClpP protease, priming neo-substrates for degradation. In addition to their targeting function, BacPROTACs activate ClpC, transforming the resting unfoldase into its functional state. The induced higher-order oligomer was visualized by cryo-EM analysis, providing a structural snapshot of activated ClpC unfolding a protein substrate. Finally, drug susceptibility and degradation assays performed in mycobacteria demonstrate in vivo activity of BacPROTACs, allowing selective targeting of endogenous proteins via fusion to an established degron. In addition to guiding antibiotic discovery, the BacPROTAC technology presents a versatile research tool enabling the inducible degradation of bacterial proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11707.map.gz emd_11707.map.gz | 8.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11707-v30.xml emd-11707-v30.xml emd-11707.xml emd-11707.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11707_fsc.xml emd_11707_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11707.png emd_11707.png | 130.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11707 http://ftp.pdbj.org/pub/emdb/structures/EMD-11707 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11707 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11707 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11707_validation.pdf.gz emd_11707_validation.pdf.gz | 367.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11707_full_validation.pdf.gz emd_11707_full_validation.pdf.gz | 367.4 KB | 表示 | |
| XML形式データ |  emd_11707_validation.xml.gz emd_11707_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_11707_validation.cif.gz emd_11707_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11707 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11707 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11707 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11707 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7abrMC  7aa4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-11068 (タイトル: Single particle Data of the activated B. subtilis ClpC arranged as a tetramer of hexamers EMPIAR-11068 (タイトル: Single particle Data of the activated B. subtilis ClpC arranged as a tetramer of hexamersData size: 4.9 TB Data #1: Unaligned Multiframe micrographs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11707.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11707.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.058 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : B. subtilis ClpC (DWB mutant) hexamer bound to a substrate polypeptide
| 全体 | 名称: B. subtilis ClpC (DWB mutant) hexamer bound to a substrate polypeptide |
|---|---|
| 要素 |
|
-超分子 #1: B. subtilis ClpC (DWB mutant) hexamer bound to a substrate polypeptide
| 超分子 | 名称: B. subtilis ClpC (DWB mutant) hexamer bound to a substrate polypeptide タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #2: Negative regulator of genetic competence ClpC/MecB
| 超分子 | 名称: Negative regulator of genetic competence ClpC/MecB / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #3: substrate polypeptide
| 超分子 | 名称: substrate polypeptide / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 組換発現 | 生物種:  |
-分子 #1: Negative regulator of genetic competence ClpC/MecB
| 分子 | 名称: Negative regulator of genetic competence ClpC/MecB / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 91.206812 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMFGRFTERA QKVLALAQEE ALRLGHNNIG TEHILLGLVR EGEGIAAKAL QALGLGSEKI QKEVESLIGR GQEMSQTIHY TPRAKKVIE LSMDEARKLG HSYVGTEHIL LGLIREGEGV AARVLNNLGV SLNKARQQVL QLLGSNETGS SAAGTNSNAN T PTLDSLAR ...文字列: MMFGRFTERA QKVLALAQEE ALRLGHNNIG TEHILLGLVR EGEGIAAKAL QALGLGSEKI QKEVESLIGR GQEMSQTIHY TPRAKKVIE LSMDEARKLG HSYVGTEHIL LGLIREGEGV AARVLNNLGV SLNKARQQVL QLLGSNETGS SAAGTNSNAN T PTLDSLAR DLTAIAKEDS LDPVIGRSKE IQRVIEVLSR RTKNNPVLIG EPGVGKTAIA EGLAQQIINN EVPEILRDKR VM TLDMGTV VAGTKYRGEF EDRLKKVMDE IRQAGNIILF IDALHTLIGA GGAEGAIDAS NILKPSLARG ELQCIGATTL DEY RKYIEK DAALERRFQP IQVDQPSVDE SIQILQGLRD RYEAHHRVSI TDDAIEAAVK LSDRYISDRF LPDKAIDLID EAGS KVRLR SFTTPPNLKE LEQKLDEVRK EKDAAVQSQE FEKAASLRDT EQRLREQVED TKKSWKEKQG QENSEVTVDD IAMVV SSWT GVPVSKIAQT ETDKLLNMEN ILHSRVIGQD EAVVAVAKAV RRARAGLKDP KRPIGSFIFL GPTGVGKTEL ARALAE SIF GDEESMIRID MSEYMEKHST SRLVGSPPGY VGYDEGGQLT EKVRRKPYSV VLLDAIEKAH PDVFNILLQV LEDGRLT DS KGRTVDFRNT ILIMTSNVGA SELKRNKYVG FNVQDETQNH KDMKDKVMGE LKRAFRPEFI NRIDEIIVFH SLEKKHLT E IVSLMSDQLT KRLKEQDLSI ELTDAAKAKV AEEGVDLEYG ARPLRRAIQK HVEDRLSEEL LRGNIHKGQH IVLDVEDGE FVVKTTAKTN LEHHHHHH |
-分子 #2: substrate polypeptide
| 分子 | 名称: substrate polypeptide / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 2.230741 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 7 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 4455 / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー