+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10492 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Vip3Aa protoxin structure | ||||||||||||||||||
 マップデータ マップデータ | Half map 2 | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Vip3Aa / toxin / protoxin / beta prism / insecticidal protein / Vip3 | ||||||||||||||||||
| 機能・相同性 | Vegetative insecticide protein 3 / Vegetative insecticide protein 3A N terminal / Carbohydrate-binding, CenC-like / Carbohydrate binding domain / hydrolase activity, acting on glycosyl bonds / Galactose-binding-like domain superfamily / Vegetative insecticidal protein 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
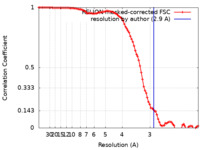
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Nunez-Ramirez R / Huesa J | ||||||||||||||||||
| 資金援助 |  スペイン, 5件 スペイン, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Molecular architecture and activation of the insecticidal protein Vip3Aa from Bacillus thuringiensis. 著者: Rafael Núñez-Ramírez / Juanjo Huesa / Yolanda Bel / Juan Ferré / Patricia Casino / Ernesto Arias-Palomo /  要旨: Bacillus thuringiensis Vip3 (Vegetative Insecticidal Protein 3) toxins are widely used in biotech crops to control Lepidopteran pests. These proteins are produced as inactive protoxins that need to ...Bacillus thuringiensis Vip3 (Vegetative Insecticidal Protein 3) toxins are widely used in biotech crops to control Lepidopteran pests. These proteins are produced as inactive protoxins that need to be activated by midgut proteases to trigger cell death. However, little is known about their three-dimensional organization and activation mechanism at the molecular level. Here, we have determined the structures of the protoxin and the protease-activated state of Vip3Aa at 2.9 Å using cryo-electron microscopy. The reconstructions show that the protoxin assembles into a pyramid-shaped tetramer with the C-terminal domains exposed to the solvent and the N-terminal region folded into a spring-loaded apex that, after protease activation, drastically remodels into an extended needle by a mechanism akin to that of influenza haemagglutinin. These results provide the molecular basis for Vip3 activation and function, and serves as a strong foundation for the development of more efficient insecticidal proteins. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10492.map.gz emd_10492.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10492-v30.xml emd-10492-v30.xml emd-10492.xml emd-10492.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10492_fsc.xml emd_10492_fsc.xml | 9.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10492.png emd_10492.png | 158.6 KB | ||
| マスクデータ |  emd_10492_msk_1.map emd_10492_msk_1.map | 70.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10492.cif.gz emd-10492.cif.gz | 6.2 KB | ||
| その他 |  emd_10492_additional_1.map.gz emd_10492_additional_1.map.gz emd_10492_additional_2.map.gz emd_10492_additional_2.map.gz emd_10492_half_map_1.map.gz emd_10492_half_map_1.map.gz emd_10492_half_map_2.map.gz emd_10492_half_map_2.map.gz | 53 MB 2.7 MB 53.3 MB 53.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10492 http://ftp.pdbj.org/pub/emdb/structures/EMD-10492 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10492 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10492 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10492_validation.pdf.gz emd_10492_validation.pdf.gz | 411.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10492_full_validation.pdf.gz emd_10492_full_validation.pdf.gz | 410.4 KB | 表示 | |
| XML形式データ |  emd_10492_validation.xml.gz emd_10492_validation.xml.gz | 15.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10492 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10492 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10492 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10492 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10492.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10492.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.048 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10492_msk_1.map emd_10492_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
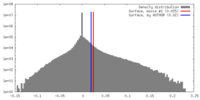
| 密度ヒストグラム |
-追加マップ: Unsharpened EM map of the Vip3Aa protoxin
| ファイル | emd_10492_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened EM map of the Vip3Aa protoxin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
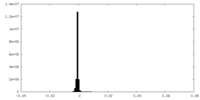
| 密度ヒストグラム |
-追加マップ: Focused classification map of the C-terminal domains of the protoxin
| ファイル | emd_10492_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused classification map of the C-terminal domains of the protoxin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10492_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
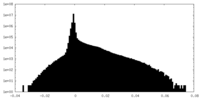
| 密度ヒストグラム |
-ハーフマップ: Focused classification map of the C-terminal domains of the protoxin
| ファイル | emd_10492_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused classification map of the C-terminal domains of the protoxin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vip3Aa protoxin
| 全体 | 名称: Vip3Aa protoxin |
|---|---|
| 要素 |
|
-超分子 #1: Vip3Aa protoxin
| 超分子 | 名称: Vip3Aa protoxin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 350 KDa |
-分子 #1: Vegetative insecticidal protein
| 分子 | 名称: Vegetative insecticidal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 88.762805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNKNNTKLST RALPSFIDYF NGIYGFATGI KDIMNMIFKT DTGGDLTLDE ILKNQQLLND ISGKLDGVNG SLNDLIAQGN LNTELSKEI LKIANEQNQV LNDVNNKLDA INTMLRVYLP KLTSMLSDVM KQNYALSLQI EYLSKQLQEI SDKLDIINVN V LINSTLTE ...文字列: MNKNNTKLST RALPSFIDYF NGIYGFATGI KDIMNMIFKT DTGGDLTLDE ILKNQQLLND ISGKLDGVNG SLNDLIAQGN LNTELSKEI LKIANEQNQV LNDVNNKLDA INTMLRVYLP KLTSMLSDVM KQNYALSLQI EYLSKQLQEI SDKLDIINVN V LINSTLTE ITPAYQRIKY VNEKFEELTF ATETSSKVKK DGSPADILDE LTELTELAKS VTKNDVDGFE FYLNTFHDVM VG NNLFGRS ALKTASELIT KENVKTSGSE VGNVYNFLIV LTALQAKAFL TLTTCRKLLG LADIDYTSIM NEHLNKEKEE FRV NILPTL SNTFSNPNYA KVKGSDEDAK MIVEAKPGHA LIGFEISNDS ITVLKVYEAK LKQNYQVDKD SLSEVIYGDM DKLL CPDQS EQIYYTNNIV FPNEYVITKI DFTKKMKTLR YEVTANFYDS STGEIDLNKK KVESSEAEYR TLSANDDGVY MPLGV ISET FLTPINGFGL QADENSRLIT LTCKSYLREL LLATDLSNKE TKLIVPPSGF ISNIVENGSI EEDNLEPWKA NNKNAY VDH TGGVNGTKAL YVHKDGGISQ FIGDKLKPKT EYVIQYTVKG KPSIHLKDEN TGYIHYEDTN NNLEDYQTIN KRFTTGT DL KGVYLILKSQ NGDEAWGDNF IILEISPSEK LLSPELINTN NWTSTGSTNI SGNTLTLYQG GRGILKQNLQ LDSFSTYR V YFSVSGDANV RIRNSREVLF EKRYMSGAKD VSEMFTTKFE KDNFYIELSQ GNNLYGGPIV HFYDVSIK UniProtKB: Vegetative insecticidal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 56.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The atomic coordinates were manually modeled de novo in the cryo-EM map using Coot, and then subjected to iterative rounds of real space refinement using Phenix |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-6tfj: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)