+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10277 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human SERINC5 in LMNG micelles, bound to Fab. | |||||||||
 マップデータ マップデータ | sharpened EM map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
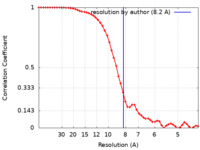
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.2 Å | |||||||||
 データ登録者 データ登録者 | Rosa A / Pye VE / Nans A / Cherepanov P | |||||||||
| 資金援助 |  米国, 米国,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: A bipartite structural organization defines the SERINC family of HIV-1 restriction factors. 著者: Valerie E Pye / Annachiara Rosa / Cinzia Bertelli / Weston B Struwe / Sarah L Maslen / Robin Corey / Idlir Liko / Mark Hassall / Giada Mattiuzzo / Allison Ballandras-Colas / Andrea Nans / ...著者: Valerie E Pye / Annachiara Rosa / Cinzia Bertelli / Weston B Struwe / Sarah L Maslen / Robin Corey / Idlir Liko / Mark Hassall / Giada Mattiuzzo / Allison Ballandras-Colas / Andrea Nans / Yasuhiro Takeuchi / Phillip J Stansfeld / J Mark Skehel / Carol V Robinson / Massimo Pizzato / Peter Cherepanov /   要旨: The human integral membrane protein SERINC5 potently restricts HIV-1 infectivity and sensitizes the virus to antibody-mediated neutralization. Here, using cryo-EM, we determine the structures of ...The human integral membrane protein SERINC5 potently restricts HIV-1 infectivity and sensitizes the virus to antibody-mediated neutralization. Here, using cryo-EM, we determine the structures of human SERINC5 and its orthologue from Drosophila melanogaster at subnanometer and near-atomic resolution, respectively. The structures reveal a novel fold comprised of ten transmembrane helices organized into two subdomains and bisected by a long diagonal helix. A lipid binding groove and clusters of conserved residues highlight potential functional sites. A structure-based mutagenesis scan identified surface-exposed regions and the interface between the subdomains of SERINC5 as critical for HIV-1-restriction activity. The same regions are also important for viral sensitization to neutralizing antibodies, directly linking the antiviral activity of SERINC5 with remodeling of the HIV-1 envelope glycoprotein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10277.map.gz emd_10277.map.gz | 9.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10277-v30.xml emd-10277-v30.xml emd-10277.xml emd-10277.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10277_fsc.xml emd_10277_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10277.png emd_10277.png | 125.3 KB | ||
| その他 |  emd_10277_additional.map.gz emd_10277_additional.map.gz emd_10277_additional_1.map.gz emd_10277_additional_1.map.gz emd_10277_half_map_1.map.gz emd_10277_half_map_1.map.gz emd_10277_half_map_2.map.gz emd_10277_half_map_2.map.gz | 7.8 MB 7.8 MB 7.9 MB 7.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10277 http://ftp.pdbj.org/pub/emdb/structures/EMD-10277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10277 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10277_validation.pdf.gz emd_10277_validation.pdf.gz | 344 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10277_full_validation.pdf.gz emd_10277_full_validation.pdf.gz | 343.1 KB | 表示 | |
| XML形式データ |  emd_10277_validation.xml.gz emd_10277_validation.xml.gz | 10.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10277 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10277 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10277.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10277.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened EM map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.18 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened EM map
| ファイル | emd_10277_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened EM map | ||||||||||||
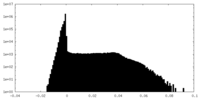
| 投影像・断面図 |
| ||||||||||||
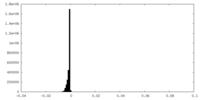
| 密度ヒストグラム |
-追加マップ: unsharpened EM map
| ファイル | emd_10277_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened EM map | ||||||||||||
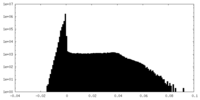
| 投影像・断面図 |
| ||||||||||||
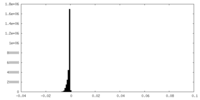
| 密度ヒストグラム |
-ハーフマップ: EM half map 2
| ファイル | emd_10277_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half map 1
| ファイル | emd_10277_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of SERINC5 with FAB bound to ECL4 in LMNG micelles
| 全体 | 名称: complex of SERINC5 with FAB bound to ECL4 in LMNG micelles |
|---|---|
| 要素 |
|
-超分子 #1: complex of SERINC5 with FAB bound to ECL4 in LMNG micelles
| 超分子 | 名称: complex of SERINC5 with FAB bound to ECL4 in LMNG micelles タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-分子 #1: SERINC5
| 分子 | 名称: SERINC5 / タイプ: protein_or_peptide / ID: 1 詳細: Full-length, wild type human SERINC5 with C-terminal TwinStrep tag. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: msaqccagql acccgsagcs lccdccprir qslstrfmya lyfilvvvlc cimmsttvah kmkehipffe dmckgikagd tceklvgysa vyrvcfgmac fffifclltl kinnskscra hihngfwffk llllgamcsg affipdqdtf lnawryvgav ggflfigiql ...文字列: msaqccagql acccgsagcs lccdccprir qslstrfmya lyfilvvvlc cimmsttvah kmkehipffe dmckgikagd tceklvgysa vyrvcfgmac fffifclltl kinnskscra hihngfwffk llllgamcsg affipdqdtf lnawryvgav ggflfigiql lllvefahkw nknwtagtas nklwyaslal vtlimysiat gglvlmavfy tqkdscmenk illgvngglc llislvaisp wvqnrqphsg llqsgviscy vtyltfsals skpaevvlde hgknvticvp dfgqdlyrde nlvtilgtsl ligcilyscl tsttrsssda lqgryaapel eiarccfcfs pggedteeqq pgkegprviy dekkgtvyiy syfhfvffla slyvmmtvtn wfnyesanie sffsgswsif wvkmascwic vllylctlva plccptrefs vgssglevlf qgpgsggsaw shpqfekggg sgggsggsaw shpqfek |
-分子 #2: FAB (antigen binding fragment from a monoclonal antibody)
| 分子 | 名称: FAB (antigen binding fragment from a monoclonal antibody) タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: amino acid sequence unknown |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 詳細: No pre-treatment | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | The complex was assembled using recombinant SERINC5 in LMNG/PS and FAB and was purified by size exclusion chromatograhy immediately prior to vitrification on holey carbon EM grids. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 7165 / 平均電子線量: 33.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -4.0 µm / 最小 デフォーカス(公称値): -1.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X