+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10272 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BamABCDE in MSP1D1 nanodisc ensemble 1-5 | |||||||||||||||
 マップデータ マップデータ | BamABCDE in MSP1D1 nanodisc ensemble 1-5 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Outer membrane / OMP / beta-barrel / folding / insertion / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane / cell outer membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
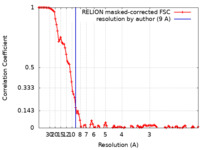
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Iadanza MG / Ranson NA | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2020 ジャーナル: Commun Biol / 年: 2020タイトル: Distortion of the bilayer and dynamics of the BAM complex in lipid nanodiscs. 著者: Matthew G Iadanza / Bob Schiffrin / Paul White / Matthew A Watson / Jim E Horne / Anna J Higgins / Antonio N Calabrese / David J Brockwell / Roman Tuma / Antreas C Kalli / Sheena E Radford / Neil A Ranson /   要旨: The β-barrel assembly machinery (BAM) catalyses the folding and insertion of β-barrel outer membrane proteins (OMPs) into the outer membranes of Gram-negative bacteria by mechanisms that remain ...The β-barrel assembly machinery (BAM) catalyses the folding and insertion of β-barrel outer membrane proteins (OMPs) into the outer membranes of Gram-negative bacteria by mechanisms that remain unclear. Here, we present an ensemble of cryoEM structures of the E. coli BamABCDE (BAM) complex in lipid nanodiscs, determined using multi-body refinement techniques. These structures, supported by single-molecule FRET measurements, describe a range of motions in the BAM complex, mostly localised within the periplasmic region of the major subunit BamA. The β-barrel domain of BamA is in a 'lateral open' conformation in all of the determined structures, suggesting that this is the most energetically favourable species in this bilayer. Strikingly, the BAM-containing lipid nanodisc is deformed, especially around BAM's lateral gate. This distortion is also captured in molecular dynamics simulations, and provides direct structural evidence for the lipid 'disruptase' activity of BAM, suggested to be an important part of its functional mechanism. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10272.map.gz emd_10272.map.gz | 8.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10272-v30.xml emd-10272-v30.xml emd-10272.xml emd-10272.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10272_fsc.xml emd_10272_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10272.png emd_10272.png | 85 KB | ||
| Filedesc metadata |  emd-10272.cif.gz emd-10272.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10272 http://ftp.pdbj.org/pub/emdb/structures/EMD-10272 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10272 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10272 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10272_validation.pdf.gz emd_10272_validation.pdf.gz | 359.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10272_full_validation.pdf.gz emd_10272_full_validation.pdf.gz | 359.3 KB | 表示 | |
| XML形式データ |  emd_10272_validation.xml.gz emd_10272_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_10272_validation.cif.gz emd_10272_validation.cif.gz | 15.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10272 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10272 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10272 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10272 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6socMC  6smxC  6sn0C  6sn2C  6sn3C  6sn4C  6sn5C  6sn7C  6sn8C  6sn9C  6so7C  6so8C  6soaC  6sobC  6sogC  6sohC  6sojC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10272.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10272.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BamABCDE in MSP1D1 nanodisc ensemble 1-5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bam complex
| 全体 | 名称: Bam complex |
|---|---|
| 要素 |
|
-超分子 #1: Bam complex
| 超分子 | 名称: Bam complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: In MSP1D1 nanodisc with E. coli polar lipid extract |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Outer membrane protein assembly factor BamA
| 分子 | 名称: Outer membrane protein assembly factor BamA / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 87.783945 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FVVKDIHFEG LQRVAVGAAL LSMPVRTGDT VNDEDISNTI RALFATGNFE DVRVLRDGDT LLVQVKERPT IASITFSGNK SVKDDMLKQ NLEASGVRVG ESLDRTTIAD IEKGLEDFYY SVGKYSASVK AVVTPLPRNR VDLKLVFQEG VSAEIQQINI V GNHAFTTD ...文字列: FVVKDIHFEG LQRVAVGAAL LSMPVRTGDT VNDEDISNTI RALFATGNFE DVRVLRDGDT LLVQVKERPT IASITFSGNK SVKDDMLKQ NLEASGVRVG ESLDRTTIAD IEKGLEDFYY SVGKYSASVK AVVTPLPRNR VDLKLVFQEG VSAEIQQINI V GNHAFTTD ELISHFQLRD EVPWWNVVGD RKYQKQKLAG DLETLRSYYL DRGYARFNID STQVSLTPDK KGIYVTVNIT EG DQYKLSG VEVSGNLAGH SAEIEQLTKI EPGELYNGTK VTKMEDDIKK LLGRYGYAYP RVQSMPEIND ADKTVKLRVN VDA GNRFYV RKIRFEGNDT SKDAVLRREM RQMEGAWLGS DLVDQGKERL NRLGFFETVD TDTQRVPGSP DQVDVVYKVK ERNT GSFNF GIGYGTESGV SFQAGVQQDN WLGTGYAVGI NGTKNDYQTY AELSVTNPYF TVDGVSLGGR LFYNDFQADD ADLSD YTNK SYGTDVTLGF PINEYNSLRA GLGYVHNSLS NMQPQVAMWR YLYSMGEHPS TSDQDNSFKT DDFTFNYGWT YNKLDR GYF PTDGSRVNLT GKVTIPGSDN EYYKVTLDTA TYVPIDDDHK WVVLGRTRWG YGDGLGGKEM PFYENFYAGG SSTVRGF QS NTIGPKAVYF PHQASNYDPD YDYECATQDG AKDLCKSDDA VGGNAMAVAS LEFITPTPFI SDKYANSVRT SFFWDMGT V WDTNWDSSQY SGYPDYSDPS NIRMSAGIAL QWMSPLGPLV FSYAQPFKKY DGDKAEQFQF NI UniProtKB: Outer membrane protein assembly factor BamA |
-分子 #2: Outer membrane protein assembly factor BamB
| 分子 | 名称: Outer membrane protein assembly factor BamB / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.692156 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LFNSEEDVVK MSPLPTVENQ FTPTTAWSTS VGSGIGNFYS NLHPALADNV VYAADRAGLV KALNADDGKE IWSVSLAEKD GWFSKEPAL LSGGVTVSGG HVYIGSEKAQ VYALNTSDGT VAWQTKVAGE ALSRPVVSDG LVLIHTSNGQ LQALNEADGA V KWTVNLDM ...文字列: LFNSEEDVVK MSPLPTVENQ FTPTTAWSTS VGSGIGNFYS NLHPALADNV VYAADRAGLV KALNADDGKE IWSVSLAEKD GWFSKEPAL LSGGVTVSGG HVYIGSEKAQ VYALNTSDGT VAWQTKVAGE ALSRPVVSDG LVLIHTSNGQ LQALNEADGA V KWTVNLDM PSLSLRGESA PTTAFGAAVV GGDNGRVSAV LMEQGQMIWQ QRISQATGST EIDRLSDVDT TPVVVNGVVF AL AYNGNLT ALDLRSGQIM WKRELGSVND FIVDGNRIYL VDQNDRVMAL TIDGGVTLWT QSDLLHRLLT SPVLYNGNLV VGD SEGYLH WINVEDGRFV AQQKVDSSGF QTEPVAADGK LLIQAKDGTV YSITR UniProtKB: UNIPROTKB: A0A2S5ZNM3 |
-分子 #3: Outer membrane protein assembly factor BamC
| 分子 | 名称: Outer membrane protein assembly factor BamC / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.096815 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: CSSDSRYKRQ VSGDEAYLEA APLAELHAPA GMILPVTSGD YAIPVTNGSG AVGKALDIR UniProtKB: UNIPROTKB: A0A4T5IPK3 |
-分子 #4: Outer membrane protein assembly factor BamD
| 分子 | 名称: Outer membrane protein assembly factor BamD / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.008967 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVPDNPPNEI YATAQQKLQD GNWRQAITQL EALDNRYPFG PYSQQVQLDL IYAYYKNADL PLAQAAIDRF IRLNPTHPNI DYVMYMRGL TNMALDDSAL QGFFGVDRSD RDPQHARAAF SDFSKLVRGY PNSQYTTDAT KRLVFLKDRL AKYEYSVAEY Y TERGAWVA ...文字列: EVPDNPPNEI YATAQQKLQD GNWRQAITQL EALDNRYPFG PYSQQVQLDL IYAYYKNADL PLAQAAIDRF IRLNPTHPNI DYVMYMRGL TNMALDDSAL QGFFGVDRSD RDPQHARAAF SDFSKLVRGY PNSQYTTDAT KRLVFLKDRL AKYEYSVAEY Y TERGAWVA VVNRVEGMLR DYPDTQATRD ALPLMENAYR QMQMNAQAEK VAKIIAANSS UniProtKB: Outer membrane protein assembly factor BamD |
-分子 #5: Outer membrane protein assembly factor BamE
| 分子 | 名称: Outer membrane protein assembly factor BamE / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.728837 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ERVVYRPDIN QGNYLTANDV SKIRVGMTQQ QVAYALGTPL MSDPFGTNTW FYVFRQQPGH EGVTQQTLTL TFNSSGVLTN IDNKPAL UniProtKB: Outer membrane protein assembly factor BamE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 281 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 4 / 実像数: 15504 / 平均露光時間: 10.0 sec. / 平均電子線量: 40.75 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.6 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)