+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10218 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
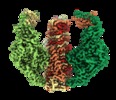
| タイトル | Structure of the Tle1 effector bound to the VgrG spike from the Type 6 secretion system | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Hydrolase / toxin / lipase / effector / bacterial | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Domain of unknown function DUF2235 / T6SS, Phospholipase effector Tle1-like, catalytic domain / Type VI secretion system spike protein VgrG2, C-terminal domain of unknown function DUF2345 / Putative type VI secretion system, Rhs element associated Vgr domain / Uncharacterized protein conserved in bacteria (DUF2345) / Putative type VI secretion system Rhs element Vgr / Type VI secretion system, RhsGE-associated Vgr family subset / Type VI secretion system, RhsGE-associated Vgr protein / Phage tail baseplate hub (GPD) / Gp5/Type VI secretion system Vgr protein, OB-fold domain ...Domain of unknown function DUF2235 / T6SS, Phospholipase effector Tle1-like, catalytic domain / Type VI secretion system spike protein VgrG2, C-terminal domain of unknown function DUF2345 / Putative type VI secretion system, Rhs element associated Vgr domain / Uncharacterized protein conserved in bacteria (DUF2345) / Putative type VI secretion system Rhs element Vgr / Type VI secretion system, RhsGE-associated Vgr family subset / Type VI secretion system, RhsGE-associated Vgr protein / Phage tail baseplate hub (GPD) / Gp5/Type VI secretion system Vgr protein, OB-fold domain / Type VI secretion system/phage-baseplate injector OB domain / Vgr protein, OB-fold domain superfamily / : 類似検索 - ドメイン・相同性 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Rapisarda C / Fronzes R | |||||||||
| 資金援助 |  フランス, フランス,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2020 ジャーナル: EMBO J / 年: 2020タイトル: Structural basis for loading and inhibition of a bacterial T6SS phospholipase effector by the VgrG spike. 著者: Nicolas Flaugnatti / Chiara Rapisarda / Martial Rey / Solène G Beauvois / Viet Anh Nguyen / Stéphane Canaan / Eric Durand / Julia Chamot-Rooke / Eric Cascales / Rémi Fronzes / Laure Journet /  要旨: The bacterial type VI secretion system (T6SS) is a macromolecular machine that injects effectors into prokaryotic and eukaryotic cells. The mode of action of the T6SS is similar to contractile phages: ...The bacterial type VI secretion system (T6SS) is a macromolecular machine that injects effectors into prokaryotic and eukaryotic cells. The mode of action of the T6SS is similar to contractile phages: the contraction of a sheath structure pushes a tube topped by a spike into target cells. Effectors are loaded onto the spike or confined into the tube. In enteroaggregative Escherichia coli, the Tle1 phospholipase binds the C-terminal extension of the VgrG trimeric spike. Here, we purify the VgrG-Tle1 complex and show that a VgrG trimer binds three Tle1 monomers and inhibits their activity. Using covalent cross-linking coupled to high-resolution mass spectrometry, we provide information on the sites of contact and further identify the requirement for a Tle1 N-terminal secretion sequence in complex formation. Finally, we report the 2.6-Å-resolution cryo-electron microscopy tri-dimensional structure of the (VgrG) -(Tle1) complex revealing how the effector binds its cargo, and how VgrG inhibits Tle1 phospholipase activity. The inhibition of Tle1 phospholipase activity once bound to VgrG suggests that Tle1 dissociation from VgrG is required upon delivery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10218.map.gz emd_10218.map.gz | 143.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10218-v30.xml emd-10218-v30.xml emd-10218.xml emd-10218.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10218.png emd_10218.png | 222 KB | ||
| Filedesc metadata |  emd-10218.cif.gz emd-10218.cif.gz | 6.4 KB | ||
| その他 |  emd_10218_additional.map.gz emd_10218_additional.map.gz emd_10218_half_map_1.map.gz emd_10218_half_map_1.map.gz emd_10218_half_map_2.map.gz emd_10218_half_map_2.map.gz | 82.5 MB 151.7 MB 151.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10218 http://ftp.pdbj.org/pub/emdb/structures/EMD-10218 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10218 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10218 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10218_validation.pdf.gz emd_10218_validation.pdf.gz | 371.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10218_full_validation.pdf.gz emd_10218_full_validation.pdf.gz | 370.3 KB | 表示 | |
| XML形式データ |  emd_10218_validation.xml.gz emd_10218_validation.xml.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10218 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10218 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10218 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10218 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10218.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10218.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.024 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
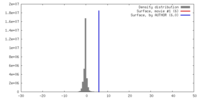
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_10218_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
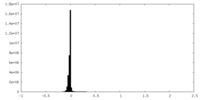
| 密度ヒストグラム |
-ハーフマップ: Half map1
| ファイル | emd_10218_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
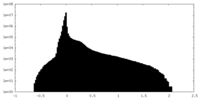
| 密度ヒストグラム |
-ハーフマップ: Half map2
| ファイル | emd_10218_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
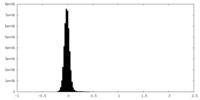
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tle1 effector bound to the VgrG spike of the type 6 secretion system
| 全体 | 名称: Tle1 effector bound to the VgrG spike of the type 6 secretion system |
|---|---|
| 要素 |
|
-超分子 #1: Tle1 effector bound to the VgrG spike of the type 6 secretion system
| 超分子 | 名称: Tle1 effector bound to the VgrG spike of the type 6 secretion system タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 956 KDa |
-分子 #1: Putative type VI secretion protein
| 分子 | 名称: Putative type VI secretion protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.657219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNLTDSLQNV LSGVGTLNRY RLDIPSCPSS LDVEDFSGTE GISKIYRYDI IFTSTDRYPD AAWFLRKSAT LIMGTGLLES LTEQKKVHG VITDFRRISG SEDQAQYRIT LKPFISLLDK QFRTHRFFVN KSVPEVVEQI LTEHGLKGWE YEFSLKRTYP K REQINQYQ ...文字列: MNLTDSLQNV LSGVGTLNRY RLDIPSCPSS LDVEDFSGTE GISKIYRYDI IFTSTDRYPD AAWFLRKSAT LIMGTGLLES LTEQKKVHG VITDFRRISG SEDQAQYRIT LKPFISLLDK QFRTHRFFVN KSVPEVVEQI LTEHGLKGWE YEFSLKRTYP K REQINQYQ ESDLRFIQRL LAEVGIFYFF TLHPDAQTEV IHFGDVQAAL IFDKTLPVNS PSGMSDSGTD SVWALNVEHR VV ESRVITN DYNHREAQNI LMSVPADMTR GEGYDTSYGE VYHYRPRHTE RGDKIDPAPE TANFWARLDH ERFLAEQTRI TGK STDASL LPAQVLTITD STPPVLPAPL QEPVLLTQLL FTGSRKSALQ VMLSAVPYSE IVCWRPPLLT RPKITGTMTA RVTS AKEGD IYAWQDASGM YRVKFDADRD DKNPGQESMP VRLAKPYSGD AYGFHFPLIQ GTEVAIAFEE GDPDRPYIAH ALHDS RHVD HVTDKNGTRN VIRTPANNKL RMEDKRGEEH IKLSTEYGGK TQLNLGHNVD ASRELRGEGA ELRTDDWISI RGGKGI FIS ADMQPQAQGK MLDMDEAIRQ LEQALSLARS MAKAATAANA TQGDISCQQR LNASLTDLTA PGMLLHAPDG IGMVSAR AL RIASGSESVG IMSGDNTDIT AGQSFTVVAE GAVSLLSRNQ GMQLLAAKGR VNIQAQSDDL SMSSQQNLDI QSSEGKVT V SANQELILAC GGAYIKLSGG NIELGCPGQI LLKSTNMQKM GPTSLDIASV EMPRGFGGGF ILTDEAGVPQ PSTPYRLTT AEGDILQGIT DENGKTAPVN TSIPSVVKVE FGKVKIHGET E UniProtKB: Putative type VI secretion protein |
-分子 #2: Putative type VI secretion protein
| 分子 | 名称: Putative type VI secretion protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 62.422754 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKYQGYDVT DATHKTSIHN DWKVVVAKKK PARGVTLTIG IFFDGTGNNR ENTASRLMKF NECSAARQGV NQKDAQSCED FLKEINKNS ISNGSYRGYY SNIHWLNILY HPDQVLKKDQ TSAQIKTYIS GIGTAAGEAD SVIGMGLGTS ILDIFEGVVT K TDEAMERI ...文字列: MTKYQGYDVT DATHKTSIHN DWKVVVAKKK PARGVTLTIG IFFDGTGNNR ENTASRLMKF NECSAARQGV NQKDAQSCED FLKEINKNS ISNGSYRGYY SNIHWLNILY HPDQVLKKDQ TSAQIKTYIS GIGTAAGEAD SVIGMGLGTS ILDIFEGVVT K TDEAMERI TQALSEFMGF NLSPDFCIAK IQFDVFGFSR GAAAARHFAN RVMEQDPAIA RAIAKGLRGD FYDGKPSGEV RF LGLFDTV AAIGGISNFF DINGRSNPGV KLELRPSVAK KVFQITAMNE YRYNFSLNSI KGMWPELALP GAHSDIGGGY NPV GSPLQE NESLFLSCPE FEIVSDDTRE MDTRVYRKAE QVRKMLMTLP ALKHILPHGK LTTKIRSIGV NNSNQRRAGV IQKQ VGAAV FFERMAVPND WANVCLRVML DAAQEAGVLF EPIRQTNTEL QLPSELIFLA DKAIAQGKAV RLGQEPQAFT EEELY IIGK YTHCSANWNI ESDGNLWVDP TTGEIFIHRF GPKGNKAFVF PNKPNDRWIR SVWYMDDQQR LNDNAVKNTK VMMSGV UniProtKB: Type VI secretion protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 53.94 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)