+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10206 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the curli secretion-assembly complex CsgG:CsgF | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Secretion Channel / Curli / Outer Membrane Protein / Nanopore Sensing / Protein Transport / Bacterial amyloid | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報curli secretion complex / curli assembly / protein secretion by the type VIII secretion system / protein transmembrane transport / single-species biofilm formation / cell outer membrane / outer membrane-bounded periplasmic space / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
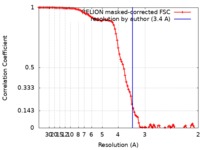
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Van der Verren SE / Remaut H | |||||||||
| 資金援助 |  ベルギー, 2件 ベルギー, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Biotechnol / 年: 2020 ジャーナル: Nat Biotechnol / 年: 2020タイトル: A dual-constriction biological nanopore resolves homonucleotide sequences with high fidelity. 著者: Sander E Van der Verren / Nani Van Gerven / Wim Jonckheere / Richard Hambley / Pratik Singh / John Kilgour / Michael Jordan / E Jayne Wallace / Lakmal Jayasinghe / Han Remaut /   要旨: Single-molecule long-read DNA sequencing with biological nanopores is fast and high-throughput but suffers reduced accuracy in homonucleotide stretches. We now combine the CsgG nanopore with the 35- ...Single-molecule long-read DNA sequencing with biological nanopores is fast and high-throughput but suffers reduced accuracy in homonucleotide stretches. We now combine the CsgG nanopore with the 35-residue N-terminal region of its extracellular interaction partner CsgF to produce a dual-constriction pore with improved signal and base-calling accuracy for homopolymer regions. The electron cryo-microscopy structure of CsgG in complex with full-length CsgF shows that the 33 N-terminal residues of CsgF bind inside the β-barrel of the pore, forming a defined second constriction. In complexes of CsgG bound to a 35-residue CsgF constriction peptide, the second constriction is separated from the primary constriction by ~25 Å. We find that both constrictions contribute to electrical signal modulation during single-stranded DNA translocation. DNA sequencing using a prototype CsgG-CsgF protein pore with two constrictions improved single-read accuracy by 25 to 70% in homopolymers up to 9 nucleotides long. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10206.map.gz emd_10206.map.gz | 10.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10206-v30.xml emd-10206-v30.xml emd-10206.xml emd-10206.xml | 14.2 KB 14.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10206_fsc.xml emd_10206_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10206.png emd_10206.png | 126.2 KB | ||
| Filedesc metadata |  emd-10206.cif.gz emd-10206.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10206 http://ftp.pdbj.org/pub/emdb/structures/EMD-10206 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10206 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10206 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10206_validation.pdf.gz emd_10206_validation.pdf.gz | 433 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10206_full_validation.pdf.gz emd_10206_full_validation.pdf.gz | 432.5 KB | 表示 | |
| XML形式データ |  emd_10206_validation.xml.gz emd_10206_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_10206_validation.cif.gz emd_10206_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10206 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10206 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10206 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10206 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10206.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10206.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CsgG:CsgF complex in DDM
| 全体 | 名称: CsgG:CsgF complex in DDM |
|---|---|
| 要素 |
|
-超分子 #1: CsgG:CsgF complex in DDM
| 超分子 | 名称: CsgG:CsgF complex in DDM / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Curli production assembly/transport component CsgF
| 分子 | 名称: Curli production assembly/transport component CsgF / タイプ: protein_or_peptide / ID: 1 / 詳細: Only first 35 residues were visible and built / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.744857 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GTMTFQFRNP NFGGNPNNGA FLLNSAQAQN SYKDPSYNDD FGIETPSALD NFTQAIQSQI LGGLLSNINT GKPGRMVTND YIVDIANRD GQLQLNVTDR KTGQTSTIQV SGLQNNSTDF HHHHHH UniProtKB: Curli production assembly/transport component CsgF |
-分子 #2: Curli production assembly/transport component CsgG
| 分子 | 名称: Curli production assembly/transport component CsgG / タイプ: protein_or_peptide / ID: 2 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.110193 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: CLTAPPKEAA RPTLMPRAQS YKDLTHLPAP TGKIFVSVYN IQDETGQFKP YPASNFSTAV PQSATAMLVT ALKDSRWFIP LERQGLQNL LNERKIIRAA QENGTVAINN RIPLQSLTAA NIMVEGSIIG YESNVKSGGV GARYFGIGAD TQYQLDQIAV N LRVVNVST ...文字列: CLTAPPKEAA RPTLMPRAQS YKDLTHLPAP TGKIFVSVYN IQDETGQFKP YPASNFSTAV PQSATAMLVT ALKDSRWFIP LERQGLQNL LNERKIIRAA QENGTVAINN RIPLQSLTAA NIMVEGSIIG YESNVKSGGV GARYFGIGAD TQYQLDQIAV N LRVVNVST GEILSSVNTS KTILSYEVQA GVFRFIDYQR LLEGEVGYTS NEPVMLCLMS AIETGVIFLI NDGIDRGLWD LQ NKAERQN DILVKYRHMS VPPESSAWSH PQFEK UniProtKB: Curli production assembly/transport component CsgG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY | ||||||||
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 2045 / 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)