+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0840 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Drosophila CTP synthase substrate-bound filament | |||||||||
 マップデータ マップデータ | the structure of CTPs from drosophila with substrate | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | substrate-bound / filament / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Interconversion of nucleotide di- and triphosphates / larval lymph gland hemopoiesis / cytoophidium / CTP synthase (glutamine hydrolysing) / CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / ATP binding / identical protein binding ...Interconversion of nucleotide di- and triphosphates / larval lymph gland hemopoiesis / cytoophidium / CTP synthase (glutamine hydrolysing) / CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / ATP binding / identical protein binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
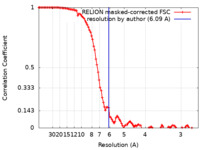
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 6.09 Å | |||||||||
 データ登録者 データ登録者 | Ji-Long L / Xian Z | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Genet Genomics / 年: 2019 ジャーナル: J Genet Genomics / 年: 2019タイトル: Drosophila CTP synthase can form distinct substrate- and product-bound filaments. 著者: Xian Zhou / Chen-Jun Guo / Huan-Huan Hu / Jiale Zhong / Qianqian Sun / Dandan Liu / Shuang Zhou / Chia Chun Chang / Ji-Long Liu /  要旨: Intracellular compartmentation is a key strategy for the functioning of a cell. In 2010, several studies revealed that the metabolic enzyme CTP synthase (CTPS) can form filamentous structures termed ...Intracellular compartmentation is a key strategy for the functioning of a cell. In 2010, several studies revealed that the metabolic enzyme CTP synthase (CTPS) can form filamentous structures termed cytoophidia in prokaryotic and eukaryotic cells. However, recent structural studies showed that CTPS only forms inactive product-bound filaments in bacteria while forming active substrate-bound filaments in eukaryotic cells. In this study, using negative staining and cryo-electron microscopy, we demonstrate that Drosophila CTPS, whether in substrate-bound or product-bound form, can form filaments. Our results challenge the previous model and indicate that substrate-bound and product-bound filaments can coexist in the same species. We speculate that the ability to switch between active and inactive cytoophidia in the same cells provides an additional layer of metabolic regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0840.map.gz emd_0840.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0840-v30.xml emd-0840-v30.xml emd-0840.xml emd-0840.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0840_fsc.xml emd_0840_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0840.png emd_0840.png | 30.2 KB | ||
| マスクデータ |  emd_0840_msk_1.map emd_0840_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-0840.cif.gz emd-0840.cif.gz | 5.4 KB | ||
| その他 |  emd_0840_half_map_1.map.gz emd_0840_half_map_1.map.gz emd_0840_half_map_2.map.gz emd_0840_half_map_2.map.gz | 6.6 MB 6.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0840 http://ftp.pdbj.org/pub/emdb/structures/EMD-0840 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0840 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0840 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0840.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0840.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | the structure of CTPs from drosophila with substrate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0840_msk_1.map emd_0840_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_0840_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_0840_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Drosophila substrate-bound CTP synthase
| 全体 | 名称: Drosophila substrate-bound CTP synthase |
|---|---|
| 要素 |
|
-超分子 #1: Drosophila substrate-bound CTP synthase
| 超分子 | 名称: Drosophila substrate-bound CTP synthase / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: CTP synthase
| 分子 | 名称: CTP synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: CTP synthase (glutamine hydrolysing) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.11848 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKYILVTGGV ISGVGKGVIA SSFGTLLKSC GLDVTSIKID PYINIDAGTF SPYEHGEVYV LDDGAEVDLD LGNYERFLDV TLHRDNNIT TGKIYKLVIE KERTGEYLGK TVQVVPHITD AIQEWVERVA QTPVQGSSKP QVCIVELGGT IGDIEGMPFV E AFRQFQFR ...文字列: MKYILVTGGV ISGVGKGVIA SSFGTLLKSC GLDVTSIKID PYINIDAGTF SPYEHGEVYV LDDGAEVDLD LGNYERFLDV TLHRDNNIT TGKIYKLVIE KERTGEYLGK TVQVVPHITD AIQEWVERVA QTPVQGSSKP QVCIVELGGT IGDIEGMPFV E AFRQFQFR VKRENFCLAH VSLVPLPKAT GEPKTKPTQS SVRELRGCGL SPDLIVCRSE KPIGLEVKEK ISNFCHVGPD QV ICIHDLN SIYHVPLLME QNGVIEYLNE RLQLNIDMSK RTKCLQQWRD LARRTETVRR EVCIAVVGKY TKFTDSYASV VKA LQHAAL AVNRKLELVF IESCLLEEET LHSEPSKYHK EWQKLCDSHG ILVPGGFGSR GMEGKIRACQ WARENQKPLL GICL GLQAA VIEFARNKLG LKDANTTEID PNTANALVID MPEHHTGQLG GTMRLGKRIT VFSDGPSVIR QLYGNPKSVQ ERHRH RYEV NPKYVHLLEE QGMRFVGTDV DKTRMEIIEL SGHPYFVATQ YHPEYLSRPL KPSPPFLGLI LASVDRLNQY IQRGCR LS UniProtKB: CTP synthase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)