+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0499 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Helicobacter pylori urea channel in open state. | |||||||||
 マップデータ マップデータ | em-volume_P1.map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicobacter pylori / urea channel / open state / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 | AmiS/UreI transporter / AmiS/UreI transporter superfamily / AmiS/UreI family transporter / identical protein binding / plasma membrane / Acid-activated urea channel 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Helicobacter pylori (strain J99 / ATCC 700824) (ピロリ菌) Helicobacter pylori (strain J99 / ATCC 700824) (ピロリ菌) | |||||||||
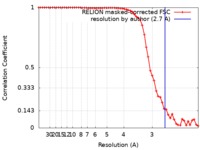
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Cui YX / Zhou K | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2019 ジャーナル: Sci Adv / 年: 2019タイトル: pH-dependent gating mechanism of the urea channel revealed by cryo-EM. 著者: Yanxiang Cui / Kang Zhou / David Strugatsky / Yi Wen / George Sachs / Z Hong Zhou / Keith Munson /  要旨: The urea channel of (UreI) is an ideal drug target for preventing gastric cancer but incomplete understanding of its gating mechanism has hampered development of inhibitors for the eradication of . ...The urea channel of (UreI) is an ideal drug target for preventing gastric cancer but incomplete understanding of its gating mechanism has hampered development of inhibitors for the eradication of . Here, we present the cryo-EM structures of UreI in closed and open conformations, both at a resolution of 2.7 Å. Our hexameric structures of this small membrane protein (~21 kDa/protomer) resolve its periplasmic loops and carboxyl terminus that close and open the channel, and define a gating mechanism that is pH dependent and requires cooperativity between protomers in the hexamer. Gating is further associated with well-resolved changes in the channel-lining residues that modify the shape and length of the urea pore. Site-specific mutations in the periplasmic domain and urea pore identified key residues important for channel function. Drugs blocking the urea pore based on our structures should lead to a new strategy for eradication. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0499.map.gz emd_0499.map.gz | 14.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0499-v30.xml emd-0499-v30.xml emd-0499.xml emd-0499.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0499_fsc.xml emd_0499_fsc.xml | 5.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0499.png emd_0499.png | 3 MB | ||
| マスクデータ |  emd_0499_msk_1.map emd_0499_msk_1.map | 15.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-0499.cif.gz emd-0499.cif.gz | 6.3 KB | ||
| その他 |  emd_0499_half_map_1.map.gz emd_0499_half_map_1.map.gz emd_0499_half_map_2.map.gz emd_0499_half_map_2.map.gz | 11.1 MB 11.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0499 http://ftp.pdbj.org/pub/emdb/structures/EMD-0499 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0499 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0499 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0499.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0499.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-volume_P1.map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
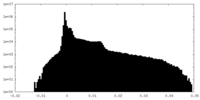
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0499_msk_1.map emd_0499_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
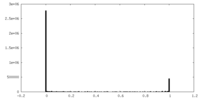
| 密度ヒストグラム |
-ハーフマップ: em-half-volume P1
| ファイル | emd_0499_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
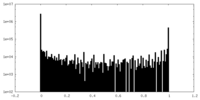
| 密度ヒストグラム |
-ハーフマップ: em-half-volume P2
| ファイル | emd_0499_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
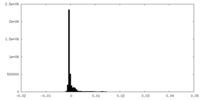
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : urea channel of Helicobacter pylori
| 全体 | 名称: urea channel of Helicobacter pylori |
|---|---|
| 要素 |
|
-超分子 #1: urea channel of Helicobacter pylori
| 超分子 | 名称: urea channel of Helicobacter pylori / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20 KDa |
-分子 #1: Acid-activated urea channel
| 分子 | 名称: Acid-activated urea channel / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Helicobacter pylori (strain J99 / ATCC 700824) (ピロリ菌) Helicobacter pylori (strain J99 / ATCC 700824) (ピロリ菌)株: J99 / ATCC 700824 |
| 分子量 | 理論値: 22.533258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLGLVLLYVG IVLISNGICG LTKVDPKSTA VMNFFVGGLS IVCNVVVITY SALHPTAPVE GHHHHHHAED IVQVSHHLTS FYGPATGLL FGFTYLYAAI NHTFGLDWRP YSWYSLFVAI NTVPAAILSH YSDMLDDHKV LGITEGDWWA IIWLAWGVLW L TAFIENIL ...文字列: MLGLVLLYVG IVLISNGICG LTKVDPKSTA VMNFFVGGLS IVCNVVVITY SALHPTAPVE GHHHHHHAED IVQVSHHLTS FYGPATGLL FGFTYLYAAI NHTFGLDWRP YSWYSLFVAI NTVPAAILSH YSDMLDDHKV LGITEGDWWA IIWLAWGVLW L TAFIENIL KIPLGKFTPW LAIIEGILTA WIPAWLLFIQ HWV UniProtKB: Acid-activated urea channel |
-分子 #2: 1,2-DIMYRISTOYL-SN-GLYCERO-3-PHOSPHATE
| 分子 | 名称: 1,2-DIMYRISTOYL-SN-GLYCERO-3-PHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 18 / 式: XP4 |
|---|---|
| 分子量 | 理論値: 591.777 Da |
| Chemical component information |  ChemComp-XP4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: FORMVAR / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 98.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 2-59 / 撮影したグリッド数: 1 / 実像数: 2901 / 平均露光時間: 9.0 sec. / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.6 µm / 最小 デフォーカス(補正後): 1.2 µm / 倍率(補正後): 46730 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)