+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0204 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | OBP chaperonin in the nucleotide-free state | |||||||||
 マップデータ マップデータ | A symmetry-free cryo-EM structure of a single-ring chaperonin encoded by gene 246 of bacteriophage OBP Pseudomonas fluorescens in the nucleotide-free state. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | chaperonin / nucleotide-free / CHAPERONE | |||||||||
| 機能・相同性 | Chaperone tailless complex polypeptide 1 (TCP-1) / GroEL-like equatorial domain superfamily / TCP-1-like chaperonin intermediate domain superfamily / GroEL-like apical domain superfamily / TCP-1/cpn60 chaperonin family / Chaperonin Cpn60/GroEL/TCP-1 family / ATP binding / Putative chaperonin GroEL 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage OBP (ファージ) Pseudomonas phage OBP (ファージ) | |||||||||
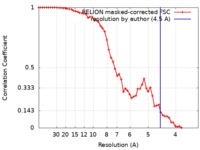
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Stanishneva-Konovalova TB / Pichkur EB / Sokolova OS | |||||||||
| 資金援助 |  ロシア, 2件 ロシア, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2020 ジャーナル: J Struct Biol / 年: 2020タイトル: Cryo-EM reveals an asymmetry in a novel single-ring viral chaperonin. 著者: Tatiana B Stanishneva-Konovalova / Pavel I Semenyuk / Lidia P Kurochkina / Evgeny B Pichkur / Alexander L Vasilyev / Mikhail V Kovalchuk / Mikhail P Kirpichnikov / Olga S Sokolova /  要旨: Chaperonins are ubiquitously present protein complexes, which assist the proper folding of newly synthesized proteins and prevent aggregation of denatured proteins in an ATP-dependent manner. They ...Chaperonins are ubiquitously present protein complexes, which assist the proper folding of newly synthesized proteins and prevent aggregation of denatured proteins in an ATP-dependent manner. They are classified into group I (bacterial, mitochondrial, chloroplast chaperonins) and group II (archaeal and eukaryotic cytosolic variants). However, both of these groups do not include recently discovered viral chaperonins. Here, we solved the symmetry-free cryo-EM structures of a single-ring chaperonin encoded by the gene 246 of bacteriophage OBP Pseudomonas fluorescens, in the nucleotide-free, ATPγS-, and ADP-bound states, with resolutions of 4.3 Å, 5.0 Å, and 6 Å, respectively. The structure of OBP chaperonin reveals a unique subunit arrangement, with three pairs of subunits and one unpaired subunit. Each pair combines subunits in two possible conformations, differing in nucleotide-binding affinity. The binding of nucleotides results in the increase of subunits' conformational variability. Due to its unique structural and functional features, OBP chaperonin can represent a new group. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0204.map.gz emd_0204.map.gz | 56.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0204-v30.xml emd-0204-v30.xml emd-0204.xml emd-0204.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0204_fsc.xml emd_0204_fsc.xml | 5.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0204.png emd_0204.png | 203.7 KB | ||
| Filedesc metadata |  emd-0204.cif.gz emd-0204.cif.gz | 5.3 KB | ||
| その他 |  emd_0204_additional.map.gz emd_0204_additional.map.gz | 9.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0204 http://ftp.pdbj.org/pub/emdb/structures/EMD-0204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0204 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0204_validation.pdf.gz emd_0204_validation.pdf.gz | 297.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0204_full_validation.pdf.gz emd_0204_full_validation.pdf.gz | 296.7 KB | 表示 | |
| XML形式データ |  emd_0204_validation.xml.gz emd_0204_validation.xml.gz | 9.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0204 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0204 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0204 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0204 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0204.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0204.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A symmetry-free cryo-EM structure of a single-ring chaperonin encoded by gene 246 of bacteriophage OBP Pseudomonas fluorescens in the nucleotide-free state. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: A symmetry-free cryo-EM structure of a single-ring chaperonin...
| ファイル | emd_0204_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A symmetry-free cryo-EM structure of a single-ring chaperonin encoded by gene 246 of bacteriophage OBP Pseudomonas fluorescens in the nucleotide-free state. | ||||||||||||
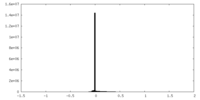
| 投影像・断面図 |
| ||||||||||||
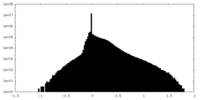
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : OBP chaperonin in the nucleotide-free state
| 全体 | 名称: OBP chaperonin in the nucleotide-free state |
|---|---|
| 要素 |
|
-超分子 #1: OBP chaperonin in the nucleotide-free state
| 超分子 | 名称: OBP chaperonin in the nucleotide-free state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage OBP (ファージ) Pseudomonas phage OBP (ファージ) |
| 分子量 | 理論値: 420 KDa |
-分子 #1: Putative chaperonin GroEL
| 分子 | 名称: Putative chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage OBP (ファージ) Pseudomonas phage OBP (ファージ) |
| 分子量 | 理論値: 57.259703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TEISFTNKND TREIVDEVVE NAFSSVCSTM GPNGNYVVIN QLNSPKVTKD GVSVARALDF NEARRNMIAK IITEPSIKTD AEVGDGTTT TVFITYHLYQ KFKDAMSFAN TRYLDTLIKQ VLQYIGTLIQ PGEIESEMFR NMLLTSSNYE EEIVDKILDI Y REHKNPNI ...文字列: TEISFTNKND TREIVDEVVE NAFSSVCSTM GPNGNYVVIN QLNSPKVTKD GVSVARALDF NEARRNMIAK IITEPSIKTD AEVGDGTTT TVFITYHLYQ KFKDAMSFAN TRYLDTLIKQ VLQYIGTLIQ PGEIESEMFR NMLLTSSNYE EEIVDKILDI Y REHKNPNI HLEKSPMLPA DEVKMTKEIY FEGSFPIETQ VPANGAYVVG PEKVGVVLID GSIRAYPTQL INALLNRFID NP VVLMARN FEPEVIAAIN NENQRLGTSR IFAYKVNAAG LLGAGTIDDL GRLLNIGPVF DVNSVDPALV KYNDVTLWLG RKG ILLDKS IEEVESRADS ILEGLDNRYE ALGIIERQTP IGRELNRRIG RLRANNVTIK VTGVTVSDAS ERWARYEDVM KAAR TGQQF GVIPGIGYGY LMASKWLEAN VPQQSDEKLE KCRIGLIEVL RAQYEHLTGH DGSAENPIFI DLVTGQESDT PMNVY DNAA ATMIALEGAW QTAKTLGKIS NVMGRSNTNY A UniProtKB: Putative chaperonin GroEL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X