+検索条件
-Structure paper
| タイトル | Cryo-EM reveals a nearly complete PCNA loading process and unique features of the human alternative clamp loader CTF18-RFC. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 121, Issue 18, Page e2319727121, Year 2024 |
| 掲載日 | 2024年4月30日 |
 著者 著者 | Qing He / Feng Wang / Michael E O'Donnell / Huilin Li /  |
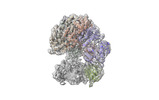
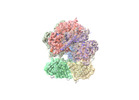
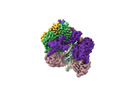
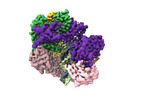
| PubMed 要旨 | The DNA sliding clamp PCNA is a multipurpose platform for DNA polymerases and many other proteins involved in DNA metabolism. The topologically closed PCNA ring needs to be cracked open and loaded ...The DNA sliding clamp PCNA is a multipurpose platform for DNA polymerases and many other proteins involved in DNA metabolism. The topologically closed PCNA ring needs to be cracked open and loaded onto DNA by a clamp loader, e.g., the well-studied pentameric ATPase complex RFC (RFC1-5). The CTF18-RFC complex is an alternative clamp loader found recently to bind the leading strand DNA polymerase ε and load PCNA onto leading strand DNA, but its structure and the loading mechanism have been unknown. By cryo-EM analysis of in vitro assembled human CTF18-RFC-DNA-PCNA complex, we have captured seven loading intermediates, revealing a detailed PCNA loading mechanism onto a 3'-ss/dsDNA junction by CTF18-RFC. Interestingly, the alternative loader has evolved a highly mobile CTF18 AAA+ module likely to lower the loading activity, perhaps to avoid competition with the RFC and to limit its role to leading strand clamp loading. To compensate for the lost stability due to the mobile AAA+ module, CTF18 has evolved a unique β-hairpin motif that reaches across RFC2 to interact with RFC5, thereby stabilizing the pentameric complex. Further, we found that CTF18 also contains a separation pin to locally melt DNA from the 3'-end of the primer; this ensures its ability to load PCNA to any 3'-ss/dsDNA junction, facilitated by the binding energy of the E-plug to the major groove. Our study reveals unique structural features of the human CTF18-RFC and contributes to a broader understanding of PCNA loading by the alternative clamp loaders. |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:38669181 / PubMed:38669181 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.75 - 3.35 Å |
| 構造データ | EMDB-42383: Cryo-EM map of the human CTF18-RFC-PCNA binary complex in the three-subunit binding state (state 2) EMDB-42384: Cryo-EM map of the human CTF18-RFC-PCNA binary complex in the four-subunit binding state (state 3) EMDB-42385: Cryo-EM map of the human CTF18-RFC-PCNA-DNA ternary complex with narrow PCNA opening state I (state 5) EMDB-42386: Cryo-EM map of the human CTF18-RFC-PCNA-DNA ternary complex in the five-subunit binding state (state 4) EMDB-42388: Cryo-EM map of the human CTF18-RFC-PCNA-DNA ternary complex with narrow PCNA opening state II (state 6) EMDB-42389: Cryo-EM map of the human CTF18-RFC-PCNA-DNA ternary complex with cracked and closed PCNA (state 7) EMDB-42406: Cryo-EM map of the human CTF18-RFC alone in the apo state (State 1) |
| 化合物 |  ChemComp-MG:  ChemComp-AGS:  ChemComp-ADP: |
| 由来 |
|
 キーワード キーワード | REPLICATION/DNA / DNA clamp loader complex / REPLICATION-DNA complex / REPLICATION |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







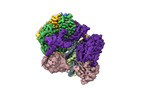

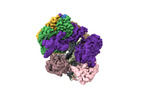



 homo sapiens (ヒト)
homo sapiens (ヒト)