+検索条件
-Structure paper
| タイトル | Interplay between an ATP-binding cassette F protein and the ribosome from Mycobacterium tuberculosis. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 13, Issue 1, Page 432, Year 2022 |
| 掲載日 | 2022年1月21日 |
 著者 著者 | Zhicheng Cui / Xiaojun Li / Joonyoung Shin / Howard Gamper / Ya-Ming Hou / James C Sacchettini / Junjie Zhang /  |
| PubMed 要旨 | EttA, energy-dependent translational throttle A, is a ribosomal factor that gates ribosome entry into the translation elongation cycle. A detailed understanding of its mechanism of action is limited ...EttA, energy-dependent translational throttle A, is a ribosomal factor that gates ribosome entry into the translation elongation cycle. A detailed understanding of its mechanism of action is limited due to the lack of high-resolution structures along its ATPase cycle. Here we present the cryo-electron microscopy (cryo-EM) structures of EttA from Mycobacterium tuberculosis (Mtb), referred to as MtbEttA, in complex with the Mtb 70S ribosome initiation complex (70SIC) at the pre-hydrolysis (ADPNP) and transition (ADP-VO) states, and the crystal structure of MtbEttA alone in the post-hydrolysis (ADP) state. We observe that MtbEttA binds the E-site of the Mtb 70SIC, remodeling the P-site tRNA and the ribosomal intersubunit bridge B7a during the ribosomal ratcheting. In return, the rotation of the 30S causes conformational changes in MtbEttA, forcing the two nucleotide-binding sites (NBSs) to alternate to engage each ADPNP in the pre-hydrolysis states, followed by complete engagements of both ADP-VO molecules in the ATP-hydrolysis transition states. In the post-hydrolysis state, the conserved ATP-hydrolysis motifs of MtbEttA dissociate from both ADP molecules, leaving two nucleotide-binding domains (NBDs) in an open conformation. These structures reveal a dynamic interplay between MtbEttA and the Mtb ribosome, providing insights into the mechanism of translational regulation by EttA-like proteins. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:35064151 / PubMed:35064151 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.71 - 3.23 Å |
| 構造データ | EMDB-23961, PDB-7msc: EMDB-23962, PDB-7msh: EMDB-23969, PDB-7msm: EMDB-23972, PDB-7msz: EMDB-23974, PDB-7mt2: EMDB-23975, PDB-7mt3: EMDB-23976, PDB-7mt7:  EMDB-23981:  PDB-7mu0: |
| 化合物 |  ChemComp-ZN:  ChemComp-MG:  ChemComp-ANP: 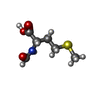 ChemComp-FME:  ChemComp-ADP: 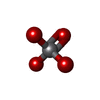 ChemComp-VO4: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / mycobacterium tuberculosis / ABCF ribosome complex / antibiotic / HYDROLASE |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について

















 mycobacterium tuberculosis h37rv (結核菌)
mycobacterium tuberculosis h37rv (結核菌)