+検索条件
-Structure paper
| タイトル | The effect of an engineered ATCUN motif on the structure and biophysical properties of the SH3 domain of c-Src tyrosine kinase. |
|---|---|
| ジャーナル・号・ページ | J. Biol. Inorg. Chem., Vol. 25, Page 621-634, Year 2020 |
| 掲載日 | 2020年1月22日 (構造データの登録日) |
 著者 著者 | Plaza-Garrido, M. / Salinas-Garcia, M.C. / Martinez, J.C. / Camara-Artigas, A. |
 リンク リンク |  J. Biol. Inorg. Chem. / J. Biol. Inorg. Chem. /  PubMed:32279137 PubMed:32279137 |
| 手法 | X線回折 |
| 解像度 | 0.9 - 1.7 Å |
| 構造データ | 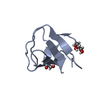 PDB-6xvm:  PDB-6xvn: 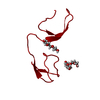 PDB-6xvo:  PDB-6xx2: 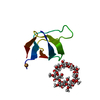 PDB-6xx3: 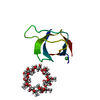 PDB-6xx4:  PDB-6xx5: |
| 化合物 |  ChemComp-GOL:  ChemComp-HOH:  ChemComp-PGE:  ChemComp-PEG:  ChemComp-CU:  ChemComp-NI: |
| 由来 |
|
 キーワード キーワード | PROTEIN BINDING / beta barrel / SH3 domain |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




