+検索条件
-Structure paper
| タイトル | Insights into the kappa / iota-carrageenan metabolism pathway of some marinePseudoalteromonasspecies. |
|---|---|
| ジャーナル・号・ページ | Commun Biol, Vol. 2, Page 474-474, Year 2019 |
| 掲載日 | 2019年7月3日 (構造データの登録日) |
 著者 著者 | Hettle, A.G. / Hobbs, J.K. / Pluvinage, B. / Vickers, C. / Abe, K.T. / Salama-Alber, O. / McGuire, B.E. / Hehemann, J.H. / Hui, J.P.M. / Berrue, F. ...Hettle, A.G. / Hobbs, J.K. / Pluvinage, B. / Vickers, C. / Abe, K.T. / Salama-Alber, O. / McGuire, B.E. / Hehemann, J.H. / Hui, J.P.M. / Berrue, F. / Banskota, A. / Zhang, J. / Bottos, E.M. / Van Hamme, J. / Boraston, A.B. |
 リンク リンク |  Commun Biol / Commun Biol /  PubMed:31886414 PubMed:31886414 |
| 手法 | X線回折 |
| 解像度 | 1.45 - 2.5 Å |
| 構造データ |  PDB-6pnu:  PDB-6pop:  PDB-6prm:  PDB-6psm:  PDB-6pso:  PDB-6pt4:  PDB-6pt6:  PDB-6pt9:  PDB-6ptk:  PDB-6ptm: |
| 化合物 |  ChemComp-EDO:  ChemComp-HOH:  ChemComp-NAP:  ChemComp-MG:  ChemComp-CA: 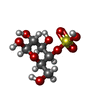 ChemComp-G4S: 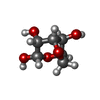 ChemComp-9RN:  ChemComp-CL:  ChemComp-ZN:  ChemComp-EPE:  ChemComp-SO4:  ChemComp-GOL: |
| 由来 |
|
 キーワード キーワード | OXIDOREDUCTASE / Dehydrogenase / HYDROLASE / S1 sulfatase / exo-carrageenase |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 pseudoalteromonas fuliginea (バクテリア)
pseudoalteromonas fuliginea (バクテリア)