+検索条件
-Structure paper
| タイトル | Human PLD structures enable drug design and characterization of isoenzyme selectivity. |
|---|---|
| ジャーナル・号・ページ | Nat. Chem. Biol., Vol. 16, Page 391-399, Year 2020 |
| 掲載日 | 2019年4月6日 (構造データの登録日) |
 著者 著者 | Metrick, C.M. / Peterson, E.A. / Santoro, J.C. / Enyedy, I.J. / Murugan, P. / Chen, T. / Michelsen, K. / Cullivan, M. / Spilker, K.A. / Kumar, P.R. ...Metrick, C.M. / Peterson, E.A. / Santoro, J.C. / Enyedy, I.J. / Murugan, P. / Chen, T. / Michelsen, K. / Cullivan, M. / Spilker, K.A. / Kumar, P.R. / May-Dracka, T.L. / Chodaparambil, J.V. |
 リンク リンク |  Nat. Chem. Biol. / Nat. Chem. Biol. /  PubMed:32042197 PubMed:32042197 |
| 手法 | X線回折 |
| 解像度 | 1.895 - 3.2 Å |
| 構造データ |  PDB-6ohm:  PDB-6oho:  PDB-6ohp:  PDB-6ohq:  PDB-6ohr:  PDB-6ohs: |
| 化合物 |  ChemComp-WO4:  ChemComp-SO4:  ChemComp-GOL:  ChemComp-HOH:  ChemComp-DTT: 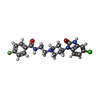 ChemComp-MJV: 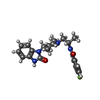 ChemComp-MKA: 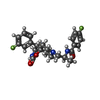 ChemComp-MKG:  ChemComp-MJY: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / phosphodiesterase / HKD motif / hydrolase/hydrolase inhibitor / hydrolase-hydrolase inhibitor complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)