+検索条件
-Structure paper
| タイトル | Ternary complex of human ROR gamma ligand-binding domain, inverse agonist and SMRT peptide shows a unique mechanism of corepressor recruitment |
|---|---|
| ジャーナル・号・ページ | Genes Cells, Vol. 22, Page 535-551, Year 2017 |
| 掲載日 | 2017年3月3日 (構造データの登録日) |
 著者 著者 | Noguchi, M. / Nomura, A. / Murase, K. / Doi, S. / Yamaguchi, K. / Hirata, K. / Shiozaki, M. / Hirashima, S. / Kotoku, M. / Yamaguchi, T. ...Noguchi, M. / Nomura, A. / Murase, K. / Doi, S. / Yamaguchi, K. / Hirata, K. / Shiozaki, M. / Hirashima, S. / Kotoku, M. / Yamaguchi, T. / Katsuda, Y. / Steensma, R. / Li, X. / Tao, H. / Tse, B. / Fenn, M. / Babine, R. / Bradley, E. / Crowe, P. / Thacher, S. / Adachi, T. / Kamada, M. |
 リンク リンク |  Genes Cells / Genes Cells /  PubMed:28493531 PubMed:28493531 |
| 手法 | X線回折 |
| 解像度 | 2 - 2.6 Å |
| 構造データ |  PDB-5x8q:  PDB-5x8s:  PDB-5x8u:  PDB-5x8w:  PDB-5x8x: |
| 化合物 | 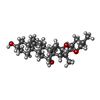 ChemComp-82R:  ChemComp-HOH: 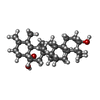 ChemComp-6Q5: 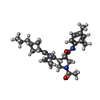 ChemComp-82O: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE/IHIBITOR / Inhibitor / Ternary Complex / Nuclear Receptor / TRANSFERASE-IHIBITOR complex / TRANSFERASE/INHIBITOR / Binary Complex / TRANSFERASE-INHIBITOR complex / TRANSCRIPTION/INHIBITOR / TRANSCRIPTION-INHIBITOR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)