タイトル Exploring the chemical space of the lysine-binding pocket of the first kringle domain of hepatocyte growth factor/scatter factor (HGF/SF) yields a new class of inhibitors of HGF/SF-MET binding. ジャーナル・号・ページ Chem Sci , Vol. 6, Page 6147-6157, Year 2015掲載日 2015年7月20日 (構造データの登録日) Sigurdardottir, A.G. / Winter, A. / Sobkowicz, A. / Fragai, M. / Chirgadze, D. / Ascher, D.B. / Blundell, T.L. / Gherardi, E. / 手法 X線回折 解像度 1.9 - 2.5 Å 構造データ PDB-5coe HEPES 手法 : X-RAY DIFFRACTION / 解像度 : 2.18 Å
PDB-5cp9 MB605 手法 : X-RAY DIFFRACTION / 解像度 : 1.9 Å
PDB-5cs1 HGF/SF 手法 : X-RAY DIFFRACTION / 解像度 : 2 Å
PDB-5cs3 (H)EPPS 手法 : X-RAY DIFFRACTION / 解像度 : 2.5 Å
PDB-5cs5 PIPES 手法 : X-RAY DIFFRACTION / 解像度 : 1.9 Å
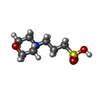
PDB-5cs9 MES 手法 : X-RAY DIFFRACTION / 解像度 : 2 Å
PDB-5csq MOPS 手法 : X-RAY DIFFRACTION / 解像度 : 1.95 Å
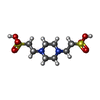
PDB-5ct1 CHES 手法 : X-RAY DIFFRACTION / 解像度 : 2 Å
PDB-5ct2 CAPS 手法 : X-RAY DIFFRACTION / 解像度 : 2 Å
PDB-5ct3 2FA 手法 : X-RAY DIFFRACTION / 解像度 : 2 Å
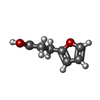
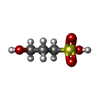
化合物 ChemComp-EPE / HEPES / pH緩衝剤*YM
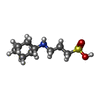
ChemComp-EP1 / EPPS / pH緩衝剤*YM
由来 homo sapiens (ヒト) / / / / / / /
 著者
著者 リンク
リンク Chem Sci /
Chem Sci /  PubMed:30090230
PubMed:30090230















 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)