+検索条件
-Structure paper
| タイトル | The impact of introducing a histidine into an apolar cavity site on docking and ligand recognition. |
|---|---|
| ジャーナル・号・ページ | J. Med. Chem., Vol. 56, Page 2874-2884, Year 2013 |
| 掲載日 | 2012年11月30日 (構造データの登録日) |
 著者 著者 | Merski, M. / Shoichet, B.K. |
 リンク リンク |  J. Med. Chem. / J. Med. Chem. /  PubMed:23473072 PubMed:23473072 |
| 手法 | X線回折 |
| 解像度 | 1.48 - 1.73 Å |
| 構造データ |  PDB-4i7j:  PDB-4i7k:  PDB-4i7l:  PDB-4i7m:  PDB-4i7n:  PDB-4i7o:  PDB-4i7p:  PDB-4i7q:  PDB-4i7r:  PDB-4i7s:  PDB-4i7t:  PDB-7sj6: |
| 化合物 |  ChemComp-BME:  ChemComp-BNZ:  ChemComp-SO4:  ChemComp-ACT:  ChemComp-HED:  ChemComp-HOH:  ChemComp-MBN:  ChemComp-IPH: 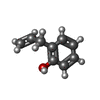 ChemComp-2LP: 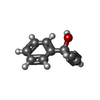 ChemComp-1DJ: 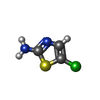 ChemComp-1DH: 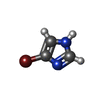 ChemComp-ES3: 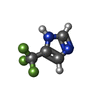 ChemComp-1DQ: 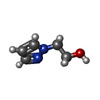 ChemComp-1DU: 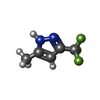 ChemComp-1DV: 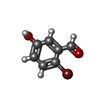 ChemComp-1DW:  ChemComp-B20: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / Organism(s): Escherichia virus T4 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 enterobacteria phage t4 (ファージ)
enterobacteria phage t4 (ファージ)