+検索条件
-Structure paper
| タイトル | Structural basis for the allosteric inhibitory mechanism of human kidney-type glutaminase (KGA) and its regulation by Raf-Mek-Erk signaling in cancer cell metabolism. |
|---|---|
| ジャーナル・号・ページ | Proc. Natl. Acad. Sci. USA, Vol. 109, Page 7705-7710, Year 2012 |
| 掲載日 | 2008年4月29日 (構造データの登録日) |
 著者 著者 | Thangavelu, K. / Pan, C.Q. / Karlberg, T. / Balaji, G. / Uttamchandani, M. / Suresh, V. / Schuler, H. / Low, B.C. / Sivaraman, J. |
 リンク リンク |  Proc. Natl. Acad. Sci. USA / Proc. Natl. Acad. Sci. USA /  PubMed:22538822 PubMed:22538822 |
| 手法 | X線回折 |
| 解像度 | 2.2 - 2.7 Å |
| 構造データ |  PDB-3czd:  PDB-3voy:  PDB-3voz:  PDB-3vp0:  PDB-3vp1:  PDB-3vp2:  PDB-3vp3:  PDB-3vp4: |
| 化合物 |  ChemComp-SO4:  ChemComp-GLU:  ChemComp-GOL:  ChemComp-HOH: 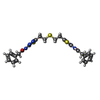 ChemComp-04A: 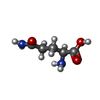 ChemComp-GLN: 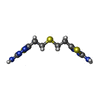 ChemComp-BP0: 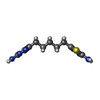 ChemComp-BP8:  ChemComp-BP9: |
| 由来 |
|
 キーワード キーワード |  HYDROLASE (加水分解酵素) / HYDROLASE (加水分解酵素) /  Glutamine (グルタミン) / Glutamine (グルタミン) /  glutamate (グルタミン酸) / kidney isoform / K-glutaminase / SGC / glutamate (グルタミン酸) / kidney isoform / K-glutaminase / SGC /  Structural Genomics Consortium / ANK repeat / Structural Genomics Consortium / ANK repeat /  Mitochondrion (ミトコンドリア) / Transit peptide / HYDROLASE/HYDROLASE INHIBTIOR / HYDROLASE-HYDROLASE INHIBTIOR complex / HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR complex Mitochondrion (ミトコンドリア) / Transit peptide / HYDROLASE/HYDROLASE INHIBTIOR / HYDROLASE-HYDROLASE INHIBTIOR complex / HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




