+検索条件
-Structure paper
| タイトル | Use of stabilizing mutations to engineer a charged group within a ligand-binding hydrophobic cavity in T4 lysozyme. |
|---|---|
| ジャーナル・号・ページ | Biochemistry, Vol. 48, Page 8842-8851, Year 2009 |
| 掲載日 | 2009年3月30日 (構造データの登録日) |
 著者 著者 | Liu, L. / Baase, W.A. / Michael, M.M. / Matthews, B.W. |
 リンク リンク |  Biochemistry / Biochemistry /  PubMed:19663503 PubMed:19663503 |
| 手法 | X線回折 |
| 解像度 | 1.45 - 2.24 Å |
| 構造データ |  PDB-3gui:  PDB-3guj:  PDB-3guk:  PDB-3gul:  PDB-3gum:  PDB-3gun:  PDB-3guo:  PDB-3gup: |
| 化合物 |  ChemComp-BME:  ChemComp-CO3:  ChemComp-TAM:  ChemComp-SO4:  ChemComp-HOH:  ChemComp-BNZ:  ChemComp-EPE:  ChemComp-MBN:  ChemComp-CA:  ChemComp-CL:  ChemComp-ACT: 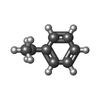 ChemComp-PYJ: 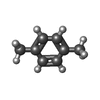 ChemComp-PXY:  ChemComp-ANL: 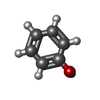 ChemComp-IPH: 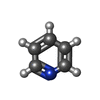 ChemComp-0PY:  ChemComp-MG: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / T4 lysozyme / apolar cavity / buried charge / ligand binding / Antimicrobial / Bacteriolytic enzyme / Glycosidase |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 enterobacteria phage t4 (ファージ)
enterobacteria phage t4 (ファージ)