+検索条件
-Structure paper
| タイトル | Analysis of flavivirus NS5 methyltransferase cap binding. |
|---|---|
| ジャーナル・号・ページ | J. Mol. Biol., Vol. 385, Page 1643-1654, Year 2009 |
| 掲載日 | 2008年10月13日 (構造データの登録日) |
 著者 著者 | Geiss, B.J. / Thompson, A.A. / Andrews, A.J. / Sons, R.L. / Gari, H.H. / Keenan, S.M. / Peersen, O.B. |
 リンク リンク |  J. Mol. Biol. / J. Mol. Biol. /  PubMed:19101564 PubMed:19101564 |
| 手法 | X線回折 |
| 解像度 | 1.45 - 2.2 Å |
| 構造データ |  PDB-3eva:  PDB-3evb:  PDB-3evc:  PDB-3evd:  PDB-3eve:  PDB-3evf:  PDB-3evg: |
| 化合物 |  ChemComp-SAH:  ChemComp-HOH:  ChemComp-GTP: 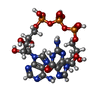 ChemComp-G3A: 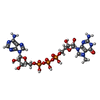 ChemComp-GTA:  ChemComp-SO4: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / yellow fever virus / NS5 methyltransferase / RNA cap binding / ATP-binding / Capsid protein / Cleavage on pair of basic residues / Endoplasmic reticulum / Envelope protein / Glycoprotein / Helicase / Hydrolase / Membrane / Metal-binding / Multifunctional enzyme / Nucleotide-binding / Nucleotidyltransferase / Nucleus / Phosphoprotein / Protease / Ribonucleoprotein / RNA replication / RNA-binding / RNA-directed RNA polymerase / Secreted / Serine protease / Transmembrane / Viral nucleoprotein / Virion / Dengue virus / Flavivirus / Transcription / Transcription regulation |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 yellow fever virus (黄熱ウイルス)
yellow fever virus (黄熱ウイルス)