+検索条件
-Structure paper
| タイトル | USP14-regulated allostery of the human proteasome by time-resolved cryo-EM. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 605, Issue 7910, Page 567-574, Year 2022 |
| 掲載日 | 2022年4月27日 |
 著者 著者 | Shuwen Zhang / Shitao Zou / Deyao Yin / Lihong Zhao / Daniel Finley / Zhaolong Wu / Youdong Mao /   |
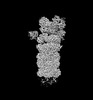
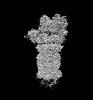
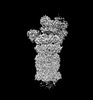
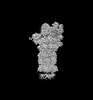
| PubMed 要旨 | Proteasomal degradation of ubiquitylated proteins is tightly regulated at multiple levels. A primary regulatory checkpoint is the removal of ubiquitin chains from substrates by the deubiquitylating ...Proteasomal degradation of ubiquitylated proteins is tightly regulated at multiple levels. A primary regulatory checkpoint is the removal of ubiquitin chains from substrates by the deubiquitylating enzyme ubiquitin-specific protease 14 (USP14), which reversibly binds the proteasome and confers the ability to edit and reject substrates. How USP14 is activated and regulates proteasome function remain unknown. Here we present high-resolution cryo-electron microscopy structures of human USP14 in complex with the 26S proteasome in 13 distinct conformational states captured during degradation of polyubiquitylated proteins. Time-resolved cryo-electron microscopy analysis of the conformational continuum revealed two parallel pathways of proteasome state transitions induced by USP14, and captured transient conversion of substrate-engaged intermediates into substrate-inhibited intermediates. On the substrate-engaged pathway, ubiquitin-dependent activation of USP14 allosterically reprograms the conformational landscape of the AAA-ATPase motor and stimulates opening of the core particle gate, enabling observation of a near-complete cycle of asymmetric ATP hydrolysis around the ATPase ring during processive substrate unfolding. Dynamic USP14-ATPase interactions decouple the ATPase activity from RPN11-catalysed deubiquitylation and kinetically introduce three regulatory checkpoints on the proteasome, at the steps of ubiquitin recognition, substrate translocation initiation and ubiquitin chain recycling. These findings provide insights into the complete functional cycle of the USP14-regulated proteasome and establish mechanistic foundations for the discovery of USP14-targeted therapies. |
 リンク リンク |  Nature / Nature /  PubMed:35477760 / PubMed:35477760 /  PubMed Central PubMed Central |
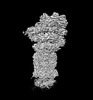
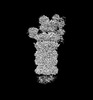
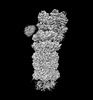
| 手法 | EM (単粒子) |
| 解像度 | 3.0 - 3.6 Å |
| 構造データ | EMDB-32272, PDB-7w37: EMDB-32273, PDB-7w38: EMDB-32274, PDB-7w39: EMDB-32275, PDB-7w3a: EMDB-32276, PDB-7w3b: EMDB-32277, PDB-7w3c: EMDB-32278, PDB-7w3f: EMDB-32279, PDB-7w3g: EMDB-32280, PDB-7w3h: EMDB-32281, PDB-7w3i: EMDB-32282, PDB-7w3j: EMDB-32283, PDB-7w3k: EMDB-32284, PDB-7w3m:  EMDB-32285: Structure of USP14-bound human 26S proteasome in state EA1_UBL with the local RPN1 density improved  EMDB-32286: Structure of USP14-bound human 26S proteasome in state EA2.0_UBL with the local RPN1 density improved  EMDB-32287: Structure of USP14-bound human 26S proteasome in state EA2.1_UBL with the local RPN1 density improved  EMDB-32288: Structure of USP14-bound human 26S proteasome in substrate-engaged state ED4_USP14 with the USP14 density improved  EMDB-32289: Structure of USP14-bound human 26S proteasome in substrate-engaged state ED0_USP14 with the RPN1 density improved  EMDB-32290: Structure of USP14-bound human 26S proteasome in substrate-engaged state ED2.0_USP14 with the RPN1 density improved  EMDB-32291: Structure of USP14-bound human 26S proteasome in substrate-engaged state ED2.1_USP14 with the USP14 density improved  EMDB-32292: Structure of USP14-bound human 26S proteasome in substrate-inhibited state SC_USP14 with the USP14 density improved |
| 化合物 |  ChemComp-ATP:  ChemComp-MG:  ChemComp-ADP:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / Proteasome / AAA-ATPase / Deubiquitinase / USP14 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



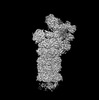



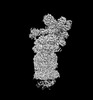

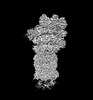

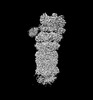










 homo sapiens (ヒト)
homo sapiens (ヒト)
