+検索条件
-Structure paper
| タイトル | Mechanisms of HIV-1 integrase resistance to dolutegravir and potent inhibition of drug-resistant variants. |
|---|---|
| ジャーナル・号・ページ | Sci Adv, Vol. 9, Issue 29, Page eadg5953, Year 2023 |
| 掲載日 | 2023年7月21日 |
 著者 著者 | Min Li / Dario Oliveira Passos / Zelin Shan / Steven J Smith / Qinfang Sun / Avik Biswas / Indrani Choudhuri / Timothy S Strutzenberg / Allan Haldane / Nanjie Deng / Zhaoyang Li / Xue Zhi Zhao / Lorenzo Briganti / Mamuka Kvaratskhelia / Terrence R Burke / Ronald M Levy / Stephen H Hughes / Robert Craigie / Dmitry Lyumkis /  |
| PubMed 要旨 | HIV-1 infection depends on the integration of viral DNA into host chromatin. Integration is mediated by the viral enzyme integrase and is blocked by integrase strand transfer inhibitors (INSTIs), ...HIV-1 infection depends on the integration of viral DNA into host chromatin. Integration is mediated by the viral enzyme integrase and is blocked by integrase strand transfer inhibitors (INSTIs), first-line antiretroviral therapeutics widely used in the clinic. Resistance to even the best INSTIs is a problem, and the mechanisms of resistance are poorly understood. Here, we analyze combinations of the mutations E138K, G140A/S, and Q148H/K/R, which confer resistance to INSTIs. The investigational drug 4d more effectively inhibited the mutants compared with the approved drug Dolutegravir (DTG). We present 11 new cryo-EM structures of drug-resistant HIV-1 intasomes bound to DTG or 4d, with better than 3-Å resolution. These structures, complemented with free energy simulations, virology, and enzymology, explain the mechanisms of DTG resistance involving E138K + G140A/S + Q148H/K/R and show why 4d maintains potency better than DTG. These data establish a foundation for further development of INSTIs that potently inhibit resistant forms in integrase. |
 リンク リンク |  Sci Adv / Sci Adv /  PubMed:37478179 PubMed:37478179 |
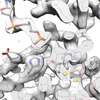
| 手法 | EM (単粒子) |
| 解像度 | 2.2 - 3.0 Å |
| 構造データ | EMDB-29307, PDB-8fn7: EMDB-29309, PDB-8fnd: EMDB-29312, PDB-8fng: EMDB-29313, PDB-8fnh: EMDB-29315, PDB-8fnj: EMDB-29317, PDB-8fnl: EMDB-29318, PDB-8fnm: EMDB-29319, PDB-8fnn: EMDB-29320, PDB-8fno: EMDB-29321, PDB-8fnp: EMDB-29322, PDB-8fnq: |
| 化合物 |  ChemComp-MG:  ChemComp-ZN: 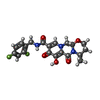 ChemComp-DLU:  ChemComp-HOH: 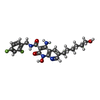 ChemComp-OZ1: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN/DNA/INHIBITOR (ウイルス性) / VIRAL PROTEIN/DNA/INHIBITOR (ウイルス性) /  Integrase (インテグラーゼ) / Integrase (インテグラーゼ) /  Nucleoprotein complex (核タンパク質) / Inhibitor / Nucleoprotein complex (核タンパク質) / Inhibitor /  Drug resistance (薬剤耐性) / Drug resistance (薬剤耐性) /  VIRAL PROTEIN-DNA-INHIBITOR complex (ウイルス性) VIRAL PROTEIN-DNA-INHIBITOR complex (ウイルス性) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について