+検索条件
-Structure paper
| タイトル | Structural characterization of NrnC identifies unifying features of dinucleotidases. |
|---|---|
| ジャーナル・号・ページ | Elife, Vol. 10, Year 2021 |
| 掲載日 | 2021年9月17日 |
 著者 著者 | Justin D Lormand / Soo-Kyoung Kim / George A Walters-Marrah / Bryce A Brownfield / J Christopher Fromme / Wade C Winkler / Jonathan R Goodson / Vincent T Lee / Holger Sondermann /   |
| PubMed 要旨 | RNA degradation is fundamental for cellular homeostasis. The process is carried out by various classes of endolytic and exolytic enzymes that together degrade an RNA polymer to mono-ribonucleotides. ...RNA degradation is fundamental for cellular homeostasis. The process is carried out by various classes of endolytic and exolytic enzymes that together degrade an RNA polymer to mono-ribonucleotides. Within the exoribonucleases, nano-RNases play a unique role as they act on the smallest breakdown products and hence catalyze the final steps in the process. We recently showed that oligoribonuclease (Orn) acts as a dedicated diribonucleotidase, defining the ultimate step in RNA degradation that is crucial for cellular fitness (Kim et al., 2019). Whether such a specific activity exists in organisms that lack Orn-type exoribonucleases remained unclear. Through quantitative structure-function analyses, we show here that NrnC-type RNases share this narrow substrate length preference with Orn. Although NrnC and Orn employ similar structural features that distinguish these two classes of dinucleotidases from other exonucleases, the key determinants for dinucleotidase activity are realized through distinct structural scaffolds. The structures, together with comparative genomic analyses of the phylogeny of DEDD-type exoribonucleases, indicate convergent evolution as the mechanism of how dinucleotidase activity emerged repeatedly in various organisms. The evolutionary pressure to maintain dinucleotidase activity further underlines the important role these analogous proteins play for cell growth. |
 リンク リンク |  Elife / Elife /  PubMed:34533457 / PubMed:34533457 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.42 - 3.69 Å |
| 構造データ | EMDB-23941, PDB-7mqb: EMDB-23942, PDB-7mqc: EMDB-23943, PDB-7mqd: EMDB-23944, PDB-7mqe: EMDB-23945, PDB-7mqf: EMDB-23946, PDB-7mqg: EMDB-23947, PDB-7mqh: EMDB-23948, PDB-7mqi:  PDB-7mpl:  PDB-7mpm:  PDB-7mpn:  PDB-7mpo:  PDB-7mpp:  PDB-7mpq:  PDB-7mpr:  PDB-7mps:  PDB-7mpt:  PDB-7mpu: |
| 化合物 |  ChemComp-HOH: 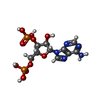 ChemComp-A3P: 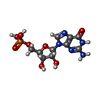 ChemComp-5GP:  ChemComp-MG:  ChemComp-MN:  ChemComp-SO4:  ChemComp-EPE:  ChemComp-PO4: |
| 由来 |
|
 キーワード キーワード | RNA BINDING PROTEIN/RNA / RNase / bacteria / enzyme / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



















 bartonella henselae (バクテリア)
bartonella henselae (バクテリア)