+検索条件
-Structure paper
| タイトル | Noncanonical assembly, neddylation and chimeric cullin-RING/RBR ubiquitylation by the 1.8 MDa CUL9 E3 ligase complex. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 31, Issue 7, Page 1083-1094, Year 2024 |
| 掲載日 | 2024年4月11日 |
 著者 著者 | Daniel Horn-Ghetko / Linus V M Hopf / Ishita Tripathi-Giesgen / Jiale Du / Sebastian Kostrhon / D Tung Vu / Viola Beier / Barbara Steigenberger / J Rajan Prabu / Luca Stier / Elias M Bruss / Matthias Mann / Yue Xiong / Brenda A Schulman /   |
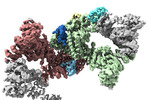
| PubMed 要旨 | Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with ...Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with E2 enzymes to catalyze ubiquitylation. However, the vertebrate-specific CUL9 complex with RBX1 (also called ROC1), of interest due to its tumor suppressive interaction with TP53, uniquely encompasses both cullin-RING and RBR domains. Here, cryo-EM, biochemistry and cellular assays elucidate a 1.8-MDa hexameric human CUL9-RBX1 assembly. Within one dimeric subcomplex, an E2-bound RBR domain is activated by neddylation of its own cullin domain and positioning from the adjacent CUL9-RBX1 in trans. Our data show CUL9 as unique among RBX1-bound cullins in dependence on the metazoan-specific UBE2F neddylation enzyme, while the RBR domain protects it from deneddylation. Substrates are recruited to various upstream domains, while ubiquitylation relies on both CUL9's neddylated cullin and RBR domains achieving self-assembled and chimeric cullin-RING/RBR E3 ligase activity. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:38605244 / PubMed:38605244 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.37 - 13.9 Å |
| 構造データ | EMDB-18214, PDB-8q7e: EMDB-18216, PDB-8q7h: 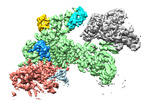 EMDB-18217: Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused on E2-like density  EMDB-18218: Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused dimeric core 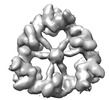 EMDB-18220: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain 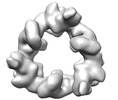 EMDB-18221: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 DOC domain 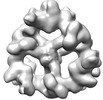 EMDB-18222: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARM9 domain 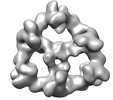 EMDB-18223: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARIH-RBR element EMDB-19179, PDB-8rhz: |
| 化合物 |  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード | LIGASE / Cullin-RING RBR E3 Ligase |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について








 homo sapiens (ヒト)
homo sapiens (ヒト)