+検索条件
-Structure paper
| タイトル | Cryo-EM structures and binding of mouse and human ACE2 to SARS-CoV-2 variants of concern indicate that mutations enabling immune escape could expand host range. |
|---|---|
| ジャーナル・号・ページ | PLoS Pathog, Vol. 19, Issue 4, Page e1011206, Year 2023 |
| 掲載日 | 2023年4月5日 |
 著者 著者 | Dongchun Ni / Priscilla Turelli / Bertrand Beckert / Sergey Nazarov / Emiko Uchikawa / Alexander Myasnikov / Florence Pojer / Didier Trono / Henning Stahlberg / Kelvin Lau /  |
| PubMed 要旨 | Investigation of potential hosts of the severe acute respiratory syndrome coronavirus-2 (SARS-CoV-2) is crucial to understanding future risks of spillover and spillback. SARS-CoV-2 has been reported ...Investigation of potential hosts of the severe acute respiratory syndrome coronavirus-2 (SARS-CoV-2) is crucial to understanding future risks of spillover and spillback. SARS-CoV-2 has been reported to be transmitted from humans to various animals after requiring relatively few mutations. There is significant interest in describing how the virus interacts with mice as they are well adapted to human environments, are used widely as infection models and can be infected. Structural and binding data of the mouse ACE2 receptor with the Spike protein of newly identified SARS-CoV-2 variants are needed to better understand the impact of immune system evading mutations present in variants of concern (VOC). Previous studies have developed mouse-adapted variants and identified residues critical for binding to heterologous ACE2 receptors. Here we report the cryo-EM structures of mouse ACE2 bound to trimeric Spike ectodomains of four different VOC: Beta, Omicron BA.1, Omicron BA.2.12.1 and Omicron BA.4/5. These variants represent the oldest to the newest variants known to bind the mouse ACE2 receptor. Our high-resolution structural data complemented with bio-layer interferometry (BLI) binding assays reveal a requirement for a combination of mutations in the Spike protein that enable binding to the mouse ACE2 receptor. |
 リンク リンク |  PLoS Pathog / PLoS Pathog /  PubMed:37018380 / PubMed:37018380 /  PubMed Central PubMed Central |
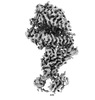
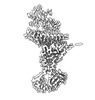
| 手法 | EM (単粒子) |
| 解像度 | 2.92 - 4.4 Å |
| 構造データ | EMDB-15588, PDB-8aqs: EMDB-15589, PDB-8aqt: EMDB-15590, PDB-8aqu: EMDB-15591, PDB-8aqv: EMDB-15592, PDB-8aqw: |
| 化合物 |  ChemComp-NAG:  ChemComp-ZN:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | VIRAL PROTEIN / SARS-COV2 / omicron / Spike / RBD / mouse / ACE2 / ANTIVIRAL PROTEIN / BA4/5 / Beta / BA.1 / BA2.12.1 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について











 homo sapiens (ヒト)
homo sapiens (ヒト)
