+検索条件
-Structure paper
| タイトル | Structural and Mechanistic Insights into Caffeine Degradation by the Bacterial N-Demethylase Complex. |
|---|---|
| ジャーナル・号・ページ | J Mol Biol, Vol. 431, Issue 19, Page 3647-3661, Year 2019 |
| 掲載日 | 2019年9月6日 |
 著者 著者 | Jun Hoe Kim / Bong Heon Kim / Shelby Brooks / Seung Yeon Kang / Ryan M Summers / Hyun Kyu Song /   |
| PubMed 要旨 | Caffeine, found in many foods, beverages, and pharmaceuticals, is the most used chemical compound for mental alertness. It is originally a natural product of plants and exists widely in environmental ...Caffeine, found in many foods, beverages, and pharmaceuticals, is the most used chemical compound for mental alertness. It is originally a natural product of plants and exists widely in environmental soil. Some bacteria, such as Pseudomonas putida CBB5, utilize caffeine as a sole carbon and nitrogen source by degrading it through sequential N-demethylation catalyzed by five enzymes (NdmA, NdmB, NdmC, NdmD, and NdmE). The environmentally friendly enzymatic reaction products, methylxanthines, are high-value biochemicals that are used in the pharmaceutical and cosmetic industries. However, the structures and biochemical properties of bacterial N-demethylases remain largely unknown. Here, we report the structures of NdmA and NdmB, the initial N- and N-specific demethylases, respectively. Reverse-oriented substrate bindings were observed in the substrate-complexed structures, offering methyl position specificity for proper N-demethylation. For efficient sequential degradation of caffeine, these enzymes form a unique heterocomplex with 3:3 stoichiometry, which was confirmed by enzymatic assays, fluorescent labeling, and small-angle x-ray scattering. The binary structure of NdmA with the ferredoxin domain of NdmD, which is the first structural information for the plant-type ferredoxin domain in a complex state, was also determined to better understand electron transport during N-demethylation. These findings broaden our understanding of the caffeine degradation mechanism by bacterial enzymes and will enable their use for industrial applications. |
 リンク リンク |  J Mol Biol / J Mol Biol /  PubMed:31412262 PubMed:31412262 |
| 手法 | SAS (X-ray synchrotron) / X線回折 |
| 解像度 | 1.65 - 2.961 Å |
| 構造データ |  SASDFD7:  SASDFE7:  SASDFF7:  SASDFG7:  SASDFH7:  SASDFJ7:  PDB-6ick:  PDB-6icl:  PDB-6icm:  PDB-6icn:  PDB-6ico:  PDB-6icp:  PDB-6icq: |
| 化合物 |  ChemComp-FES:  ChemComp-FE:  ChemComp-HOH:  ChemComp-PG4:  ChemComp-CO: 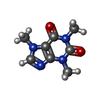 ChemComp-CFF: 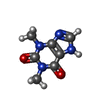 ChemComp-TEP: 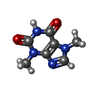 ChemComp-37T: |
| 由来 |
|
 キーワード キーワード | METAL BINDING PROTEIN / N-demethylase / Rieske oxygenase / non-heme iron center / caffeine degradation / METAL BINDING PROTEIN/OXIDOREDUCTASE / reductase plant type ferredoxin / METAL BINDING PROTEIN-OXIDOREDUCTASE complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 pseudomonas putida (バクテリア)
pseudomonas putida (バクテリア)