+検索条件
-Structure paper
| タイトル | Quantitative Conformational Analysis of Functionally Important Electrostatic Interactions in the Intrinsically Disordered Region of Delta Subunit of Bacterial RNA Polymerase. |
|---|---|
| ジャーナル・号・ページ | J Am Chem Soc, Vol. 141, Issue 42, Page 16817-16828, Year 2019 |
| 掲載日 | 2019年10月23日 |
 著者 著者 | Vojtěch Kubáň / Pavel Srb / Hana Štégnerová / Petr Padrta / Milan Zachrdla / Zuzana Jaseňáková / Hana Šanderová / Dragana Vítovská / Libor Krásný / Tomáš Koval' / Jan Dohnálek / Joanna Ziemska-Legiecka / Marcin Grynberg / Patryk Jarnot / Aleksandra Gruca / Malene Ringkjøbing Jensen / Martin Blackledge / Lukáš Žídek /    |
| PubMed 要旨 | Electrostatic interactions play important roles in the functional mechanisms exploited by intrinsically disordered proteins (IDPs). The atomic resolution description of long-range and local ...Electrostatic interactions play important roles in the functional mechanisms exploited by intrinsically disordered proteins (IDPs). The atomic resolution description of long-range and local structural propensities that can both be crucial for the function of highly charged IDPs presents significant experimental challenges. Here, we investigate the conformational behavior of the δ subunit of RNA polymerase from whose unfolded domain is highly charged, with 7 positively charged amino acids followed by 51 acidic amino acids. Using a specifically designed analytical strategy, we identify transient contacts between the two regions using a combination of NMR paramagnetic relaxation enhancements, residual dipolar couplings (RDCs), chemical shifts, and small-angle scattering. This strategy allows the resolution of long-range and local ensemble averaged structural contributions to the experimental RDCs, and reveals that the negatively charged segment folds back onto the positively charged strand, compacting the conformational sampling of the protein while remaining highly flexible in solution. Mutation of the positively charged region abrogates the long-range contact, leaving the disordered domain in an extended conformation, possibly due to local repulsion of like-charges along the chain. Remarkably, in vitro studies show that this mutation also has a significant effect on transcription activity, and results in diminished cell fitness of the mutated bacteria in vivo. This study highlights the importance of accurately describing electrostatic interactions for understanding the functional mechanisms of IDPs. |
 リンク リンク |  J Am Chem Soc / J Am Chem Soc /  PubMed:31550880 PubMed:31550880 |
| 手法 | SAS (X-ray synchrotron) |
| 構造データ | 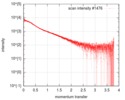 SASDF58: Delta subunit of RNA polymerase, RNAP (B. subtilis), 10mM NaCl 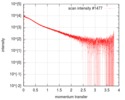 SASDF68: Delta subunit of RNA polymerase, RNAP (B. subtilis), 200 mM NaCl 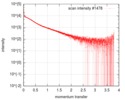 SASDF78: Delta subunit of RNA polymerase, RNAP (B. subtilis), 400 mM NaCl 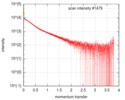 SASDF88: Delta subunit of RNA polymerase, RNAP (B. subtilis), 800 mM NaCl 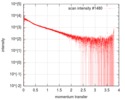 SASDF98: Delta subunit of RNA polymerase, RNAP (B. subtilis): Lysine to glutamate mutant, 10mM NaCl 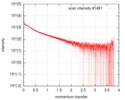 SASDFA8: Delta subunit of RNA polymerase, RNAP (B. subtilis): Lysine to glutamate mutant, 200mM NaCl  SASDFB8: Delta subunit of RNA polymerase, RNAP (B. subtilis): Lysine to glutamate mutant, 400mM NaCl 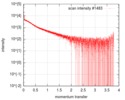 SASDFC8: Delta subunit of RNA polymerase, RNAP (B. subtilis): Lysine to glutamate mutant, 800mM NaCl |
| 由来 |
|
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




