+検索条件
-Structure paper
| タイトル | Ribosomal protein RPL39L is an efficiency factor in the cotranslational folding of a subset of proteins with alpha helical domains. |
|---|---|
| ジャーナル・号・ページ | Nucleic Acids Res, Vol. 52, Issue 15, Page 9028-9048, Year 2024 |
| 掲載日 | 2024年8月27日 |
 著者 著者 | Arka Banerjee / Meric Ataman / Maciej Jerzy Smialek / Debdatto Mookherjee / Julius Rabl / Aleksei Mironov / Lea Mues / Ludovic Enkler / Mairene Coto-Llerena / Alexander Schmidt / Daniel Boehringer / Salvatore Piscuoglio / Anne Spang / Nitish Mittal / Mihaela Zavolan /     |
| PubMed 要旨 | Increasingly many studies reveal how ribosome composition can be tuned to optimally translate the transcriptome of individual cell types. In this study, we investigated the expression pattern, ...Increasingly many studies reveal how ribosome composition can be tuned to optimally translate the transcriptome of individual cell types. In this study, we investigated the expression pattern, structure within the ribosome and effect on protein synthesis of the ribosomal protein paralog 39L (RPL39L). With a novel mass spectrometric approach we revealed the expression of RPL39L protein beyond mouse germ cells, in human pluripotent cells, cancer cell lines and tissue samples. We generated RPL39L knock-out mouse embryonic stem cell (mESC) lines and demonstrated that RPL39L impacts the dynamics of translation, to support the pluripotency and differentiation, spontaneous and along the germ cell lineage. Most differences in protein abundance between WT and RPL39L KO lines were explained by widespread autophagy. By CryoEM analysis of purified RPL39 and RPL39L-containing ribosomes we found that, unlike RPL39, RPL39L has two distinct conformations in the exposed segment of the nascent peptide exit tunnel, creating a distinct hydrophobic patch that has been predicted to support the efficient co-translational folding of alpha helices. Our study shows that ribosomal protein paralogs provide switchable modular components that can tune translation to the protein production needs of individual cell types. |
 リンク リンク |  Nucleic Acids Res / Nucleic Acids Res /  PubMed:39041433 / PubMed:39041433 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.15 - 2.66 Å |
| 構造データ | EMDB-17549, PDB-8p8m: EMDB-17550, PDB-8p8n: EMDB-17552, PDB-8p8u: EMDB-17653, PDB-8pfr: |
| 化合物 |  ChemComp-MG: 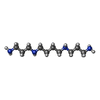 ChemComp-SPM:  ChemComp-SPD:  ChemComp-ZN:  ChemComp-CL:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / 60S ribosomal subunit / protein exit tunnel / RPL39 / RPL39L / ribosomal protein |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について













