+検索条件
-Structure paper
| タイトル | Structural basis for itraconazole-mediated NPC1 inhibition. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 11, Issue 1, Page 152, Year 2020 |
| 掲載日 | 2020年1月9日 |
 著者 著者 | Tao Long / Xiaofeng Qi / Abdirahman Hassan / Qiren Liang / Jef K De Brabander / Xiaochun Li /  |
| PubMed 要旨 | Niemann-Pick C1 (NPC1), a lysosomal protein of 13 transmembrane helices (TMs) and three lumenal domains, exports low-density-lipoprotein (LDL)-derived cholesterol from lysosomes. TMs 3-7 of NPC1 ...Niemann-Pick C1 (NPC1), a lysosomal protein of 13 transmembrane helices (TMs) and three lumenal domains, exports low-density-lipoprotein (LDL)-derived cholesterol from lysosomes. TMs 3-7 of NPC1 comprise the Sterol-Sensing Domain (SSD). Previous studies suggest that mutation of the NPC1-SSD or the addition of the anti-fungal drug itraconazole abolishes NPC1 activity in cells. However, the itraconazole binding site and the mechanism of NPC1-mediated cholesterol transport remain unknown. Here, we report a cryo-EM structure of human NPC1 bound to itraconazole, which reveals how this binding site in the center of NPC1 blocks a putative lumenal tunnel linked to the SSD. Functional assays confirm that blocking this tunnel abolishes NPC1-mediated cholesterol egress. Intriguingly, the palmitate anchor of Hedgehog occupies a similar site in the homologous tunnel of Patched, suggesting a conserved mechanism for sterol transport in this family of proteins and establishing a central function of their SSDs. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:31919352 / PubMed:31919352 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 4.02 Å |
| 構造データ | EMDB-20834, PDB-6uox: |
| 化合物 |  ChemComp-NAG: 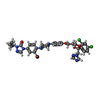 ChemComp-QDG: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / Niemann-Pick C disease / cholesterol transport / sterol-sensing domain |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





 homo sapiens (ヒト)
homo sapiens (ヒト)