| タイトル | Substrate recognition and mechanism revealed by ligand-bound polyphosphate kinase 2 structures. |
|---|
| ジャーナル・号・ページ | Proc. Natl. Acad. Sci. U.S.A., Vol. 115, Page 3350-3355, Year 2018 |
|---|
| 掲載日 | 2016年6月20日 (構造データの登録日) |
|---|
 著者 著者 | Parnell, A.E. / Mordhorst, S. / Kemper, F. / Giurrandino, M. / Prince, J.P. / Schwarzer, N.J. / Hofer, A. / Wohlwend, D. / Jessen, H.J. / Gerhardt, S. ...Parnell, A.E. / Mordhorst, S. / Kemper, F. / Giurrandino, M. / Prince, J.P. / Schwarzer, N.J. / Hofer, A. / Wohlwend, D. / Jessen, H.J. / Gerhardt, S. / Einsle, O. / Oyston, P.C.F. / Andexer, J.N. / Roach, P.L. |
|---|
 リンク リンク |  Proc. Natl. Acad. Sci. U.S.A. / Proc. Natl. Acad. Sci. U.S.A. /  PubMed:29531036 PubMed:29531036 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.903 - 2.903 Å |
|---|
| 構造データ | PDB-5lc9:
Structure of Polyphosphate Kinase from Meiothermus ruber Apo-form
手法: X-RAY DIFFRACTION / 解像度: 1.903 Å PDB-5lcd:
Structure of Polyphosphate Kinase from Meiothermus ruber bound to AMP
手法: X-RAY DIFFRACTION / 解像度: 2.66 Å PDB-5ld1:
Crystal Structure of Polyphosphate Kinase from Meiothermus ruber bound to ATP
手法: X-RAY DIFFRACTION / 解像度: 2.09 Å PDB-5ldb:
Crystal Structure of Polyphosphate Kinase from Meiothermus ruber bound to ADP
手法: X-RAY DIFFRACTION / 解像度: 2.3 Å PDB-5ll0:
Structure of Polyphosphate Kinase 2 from Francisella tularensis SCHU S4 with polyphosphate
手法: X-RAY DIFFRACTION / 解像度: 1.96 Å PDB-5llb:
Structure of Polyphosphate Kinase 2 from Francisella tularensis with AMPPCH2PPP and polyphosphate
手法: X-RAY DIFFRACTION / 解像度: 1.92 Å PDB-5llf:
Structure of Polyphosphate Kinase 2 mutant D117N from Francisella tularensis with polyphosphate
手法: X-RAY DIFFRACTION / 解像度: 2.31 Å PDB-5maq:
Crystal Structure of Polyphosphate Kinase from Meiothermus ruber bound to ADP and PPi
手法: X-RAY DIFFRACTION / 解像度: 2.46 Å PDB-5o6k:
Structure of Polyphosphate Kinase from Meiothermus ruber N121D
手法: X-RAY DIFFRACTION / 解像度: 2.903 Å PDB-5o6m:
Structure of Polyphosphate Kinase from Meiothermus ruber N121D bound to ATP
手法: X-RAY DIFFRACTION / 解像度: 2.3 Å |
|---|
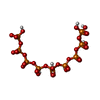
| 化合物 | ChemComp-9PI:
bis[oxidanyl-[oxidanyl-[oxidanyl(phosphonooxy)phosphoryl]oxy-phosphoryl]oxy-phosphoryl] hydrogen phosphate
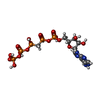
ChemComp-6YZ:
[[(2~{R},3~{S},4~{R},5~{R})-5-(6-aminopurin-9-yl)-3,4-bis(oxidanyl)oxolan-2-yl]methoxy-oxidanyl-phosphoryl]oxy-[[oxidanyl-[oxidanyl(phosphonooxy)phosphoryl]oxy-phosphoryl]methyl]phosphinic acid
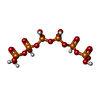
ChemComp-6YW:
[oxidanyl-[oxidanyl-[oxidanyl(phosphonooxy)phosphoryl]oxy-phosphoryl]oxy-phosphoryl] phosphono hydrogen phosphate
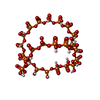
ChemComp-6YX:
[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl(phosphonooxy)phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl] phosphono hydrogen phosphate
ChemComp-6YY:
bis[oxidanyl-[oxidanyl-[oxidanyl-[oxidanyl(phosphonooxy)phosphoryl]oxy-phosphoryl]oxy-phosphoryl]oxy-phosphoryl] hydrogen phosphate
|
|---|
| 由来 |  meiothermus ruber h328 (バクテリア) meiothermus ruber h328 (バクテリア) meiothermus ruber (バクテリア) meiothermus ruber (バクテリア) francisella tularensis subsp. tularensis (strain schu s4 / schu 4) (バクテリア) francisella tularensis subsp. tularensis (strain schu s4 / schu 4) (バクテリア)
|
|---|
 キーワード キーワード | TRANSFERASE / Polyphosphate Kinase / Polyphosphate metabolism and nucleotide metabolism / Polyphosphate Kinase 2 enzyme / Complex with non-hydrolysable ATP analogue. Polyphosphate metabolism. Nucleotide metabolism / polyphosphate metabolism / nucleotide metabolism / Polyphosphate Kinase type 2 class III |
|---|
 著者
著者 リンク
リンク Proc. Natl. Acad. Sci. U.S.A. /
Proc. Natl. Acad. Sci. U.S.A. /  PubMed:29531036
PubMed:29531036


















 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 meiothermus ruber h328 (バクテリア)
meiothermus ruber h328 (バクテリア)