+検索条件
-Structure paper
| タイトル | Structural and Functional Survey of Environmental Aminoglycoside Acetyltransferases Reveals Functionality of Resistance Enzymes. |
|---|---|
| ジャーナル・号・ページ | ACS Infect Dis, Vol. 3, Page 653-665, Year 2017 |
| 掲載日 | 2015年12月3日 (構造データの登録日) |
 著者 著者 | Xu, Z. / Stogios, P.J. / Quaile, A.T. / Forsberg, K.J. / Patel, S. / Skarina, T. / Houliston, S. / Arrowsmith, C. / Dantas, G. / Savchenko, A. |
 リンク リンク |  ACS Infect Dis / ACS Infect Dis /  PubMed:28756664 PubMed:28756664 |
| 手法 | X線回折 |
| 解像度 | 1.497 - 2.15 Å |
| 構造データ |  PDB-5f46:  PDB-5f47:  PDB-5f48:  PDB-5f49:  PDB-5u08: |
| 化合物 |  ChemComp-CL:  ChemComp-HOH:  ChemComp-CA:  ChemComp-MG:  ChemComp-COA:  ChemComp-MLC: 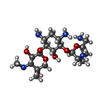 ChemComp-SIS:  ChemComp-ACT: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / GNAT fold / GCN5-N-acetyltransferase fold / acetyltransferase / aminoglycoside / antibiotic resistance / metagenome / soil / coenzyme A / Structural Genomics / Center for Structural Genomics of Infectious Diseases / CSGID / TRANSFERASE/ANTIBIOTIC / sisomicin / TRANSFERASE-ANTIBIOTIC complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 uncultured bacterium (環境試料)
uncultured bacterium (環境試料)